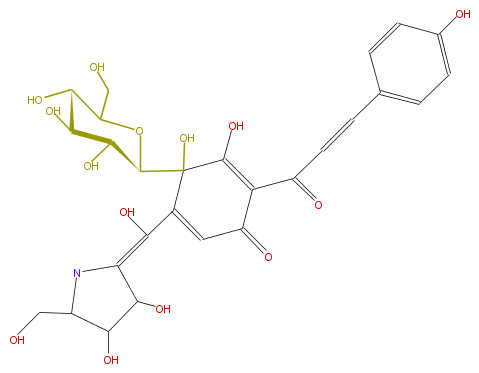
Mol:FL1CQUCCN001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 45 0 0 0 0 0 0 0 0999 V2000 | + | 42 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3910 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3910 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3910 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3910 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6765 -1.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6765 -1.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0380 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0380 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0380 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0380 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6765 -0.3253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6765 -0.3253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6765 -2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6765 -2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1385 2.3966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1385 2.3966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7197 1.8107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7197 1.8107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3082 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3082 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3188 0.2701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3188 0.2701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7374 0.8559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7374 0.8559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1488 1.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1488 1.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6248 1.8767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6248 1.8767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4835 2.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4835 2.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7316 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7316 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7197 2.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7197 2.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9708 1.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9708 1.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2045 0.1467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2045 0.1467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6617 -0.3777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6617 -0.3777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7720 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7720 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7720 -2.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7720 -2.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5675 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5675 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4359 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4359 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2031 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2031 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6156 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6156 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4406 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4406 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8531 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8531 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4406 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4406 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6156 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6156 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4615 -1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4615 -1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1122 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1122 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1122 -0.0595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1122 -0.0595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9535 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9535 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6316 -0.2679 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6316 -0.2679 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2881 -0.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2881 -0.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0157 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0157 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1909 -1.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1909 -1.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4091 -1.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4091 -1.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9479 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9479 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7334 -0.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7334 -0.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4615 -0.3223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4615 -0.3223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 2 0 0 0 0 | + | 3 7 2 0 0 0 0 |
| − | 8 9 1 1 0 0 0 | + | 8 9 1 1 0 0 0 |
| − | 9 10 1 1 0 0 0 | + | 9 10 1 1 0 0 0 |
| − | 11 10 1 1 0 0 0 | + | 11 10 1 1 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 8 1 0 0 0 0 | + | 13 8 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 8 16 1 0 0 0 0 | + | 8 16 1 0 0 0 0 |
| − | 9 17 1 0 0 0 0 | + | 9 17 1 0 0 0 0 |
| − | 10 18 1 0 0 0 0 | + | 10 18 1 0 0 0 0 |
| − | 11 6 1 0 0 0 0 | + | 11 6 1 0 0 0 0 |
| − | 6 19 1 0 0 0 0 | + | 6 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 1 32 1 0 0 0 0 | + | 1 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 32 34 2 0 0 0 0 | + | 32 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 34 1 0 0 0 0 | + | 38 34 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CQUCCN001 | + | ID FL1CQUCCN001 |
| − | KNApSAcK_ID C00014590 | + | KNApSAcK_ID C00014590 |
| − | NAME Tinctormine | + | NAME Tinctormine |
| − | CAS_RN 149475-43-4 | + | CAS_RN 149475-43-4 |
| − | FORMULA C27H31NO14 | + | FORMULA C27H31NO14 |
| − | EXACTMASS 593.174454705 | + | EXACTMASS 593.174454705 |
| − | AVERAGEMASS 593.5333800000001 | + | AVERAGEMASS 593.5333800000001 |
| − | SMILES C(C1O)(C(C(O)=3)(C(=CC(C3C(=O)C=Cc(c4)ccc(O)c4)=O)C(=C(N2)C(C(O)C2CO)O)O)O)OC(C(C1O)O)CO | + | SMILES C(C1O)(C(C(O)=3)(C(=CC(C3C(=O)C=Cc(c4)ccc(O)c4)=O)C(=C(N2)C(C(O)C2CO)O)O)O)OC(C(C1O)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 45 0 0 0 0 0 0 0 0999 V2000
-1.3910 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3910 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6765 -1.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0380 -1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0380 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6765 -0.3253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6765 -2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1385 2.3966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7197 1.8107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3082 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3188 0.2701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7374 0.8559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1488 1.5714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6248 1.8767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4835 2.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7316 2.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7197 2.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9708 1.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2045 0.1467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6617 -0.3777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7720 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7720 -2.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5675 -1.9866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4359 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2031 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6156 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4406 -2.6989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8531 -1.9844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4406 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6156 -1.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4615 -1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1122 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1122 -0.0595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9535 -0.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6316 -0.2679 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-4.2881 -0.7676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0157 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1909 -1.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4091 -1.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9479 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7334 -0.3223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4615 -0.3223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 1 0 0 0 0
3 7 2 0 0 0 0
8 9 1 1 0 0 0
9 10 1 1 0 0 0
11 10 1 1 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 8 1 0 0 0 0
13 14 1 0 0 0 0
14 15 1 0 0 0 0
8 16 1 0 0 0 0
9 17 1 0 0 0 0
10 18 1 0 0 0 0
11 6 1 0 0 0 0
6 19 1 0 0 0 0
5 20 1 0 0 0 0
4 21 1 0 0 0 0
21 22 2 0 0 0 0
21 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
28 31 1 0 0 0 0
1 32 1 0 0 0 0
32 33 1 0 0 0 0
32 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 34 1 0 0 0 0
37 39 1 0 0 0 0
38 40 1 0 0 0 0
36 41 1 0 0 0 0
41 42 1 0 0 0 0
S SKP 8
ID FL1CQUCCN001
KNApSAcK_ID C00014590
NAME Tinctormine
CAS_RN 149475-43-4
FORMULA C27H31NO14
EXACTMASS 593.174454705
AVERAGEMASS 593.5333800000001
SMILES C(C1O)(C(C(O)=3)(C(=CC(C3C(=O)C=Cc(c4)ccc(O)c4)=O)C(=C(N2)C(C(O)C2CO)O)O)O)OC(C(C1O)O)CO
M END