Mol:FL1CELNS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 33 0 0 0 0 0 0 0 0999 V2000 | + | 32 33 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6887 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6887 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6887 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6887 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1324 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1324 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5761 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5761 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5761 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5761 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1324 0.6396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1324 0.6396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0198 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0198 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4635 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4635 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0926 -0.6450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0926 -0.6450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6487 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6487 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2097 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2097 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7707 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7707 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7707 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7707 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2097 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2097 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6487 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6487 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0198 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0198 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6367 -0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6367 -0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5027 -1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5027 -1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5689 -0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5689 -0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8544 -1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8544 -1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7101 0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7101 0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1559 1.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1559 1.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6367 0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6367 0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5027 1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5027 1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7756 -0.8097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7756 -0.8097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4901 -1.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4901 -1.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0460 0.9372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0460 0.9372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5461 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5461 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8544 1.2127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8544 1.2127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4180 2.1124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4180 2.1124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4901 1.2222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4901 1.2222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9288 2.1208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9288 2.1208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 12 17 1 0 0 0 0 | + | 12 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 11 25 1 0 0 0 0 | + | 11 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 6 29 1 0 0 0 0 | + | 6 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 8 SUP | + | M STY 1 8 SUP |
| − | M SLB 1 8 8 | + | M SLB 1 8 8 |
| − | M SAL 8 2 31 32 | + | M SAL 8 2 31 32 |
| − | M SBL 8 1 32 | + | M SBL 8 1 32 |
| − | M SMT 8 OCH3 | + | M SMT 8 OCH3 |
| − | M SVB 8 32 1.4901 1.2222 | + | M SVB 8 32 1.4901 1.2222 |
| − | M STY 1 7 SUP | + | M STY 1 7 SUP |
| − | M SLB 1 7 7 | + | M SLB 1 7 7 |
| − | M SAL 7 2 29 30 | + | M SAL 7 2 29 30 |
| − | M SBL 7 1 30 | + | M SBL 7 1 30 |
| − | M SMT 7 OCH3 | + | M SMT 7 OCH3 |
| − | M SVB 7 30 -1.8544 1.2127 | + | M SVB 7 30 -1.8544 1.2127 |
| − | M STY 1 6 SUP | + | M STY 1 6 SUP |
| − | M SLB 1 6 6 | + | M SLB 1 6 6 |
| − | M SAL 6 2 27 28 | + | M SAL 6 2 27 28 |
| − | M SBL 6 1 28 | + | M SBL 6 1 28 |
| − | M SMT 6 OCH3 | + | M SMT 6 OCH3 |
| − | M SVB 6 28 -3.046 0.9372 | + | M SVB 6 28 -3.046 0.9372 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 25 26 | + | M SAL 5 2 25 26 |
| − | M SBL 5 1 26 | + | M SBL 5 1 26 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 26 0.7756 -0.8097 | + | M SVB 5 26 0.7756 -0.8097 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 23 24 | + | M SAL 4 2 23 24 |
| − | M SBL 4 1 24 | + | M SBL 4 1 24 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 24 2.3315 0.6477 | + | M SVB 4 24 2.3315 0.6477 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 21 22 | + | M SAL 3 2 21 22 |
| − | M SBL 3 1 22 | + | M SBL 3 1 22 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 22 -1.02 0.6395 | + | M SVB 3 22 -1.02 0.6395 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 19 20 | + | M SAL 2 2 19 20 |
| − | M SBL 2 1 20 | + | M SBL 2 1 20 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 20 -2.5689 -0.8056 | + | M SVB 2 20 -2.5689 -0.8056 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 17 18 | + | M SAL 1 2 17 18 |
| − | M SBL 1 1 18 | + | M SBL 1 1 18 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 18 1.9743 -0.5301 | + | M SVB 1 18 1.9743 -0.5301 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CELNS0002 | + | ID FL1CELNS0002 |
| − | KNApSAcK_ID C00006992 | + | KNApSAcK_ID C00006992 |
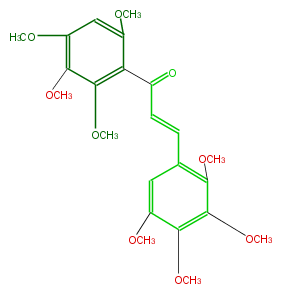
| − | NAME 2,3,4,5,2',3',4',6'-Octamethoxychalcone | + | NAME 2,3,4,5,2',3',4',6'-Octamethoxychalcone |
| − | CAS_RN 121697-04-9 | + | CAS_RN 121697-04-9 |
| − | FORMULA C23H28O9 | + | FORMULA C23H28O9 |
| − | EXACTMASS 448.17333249399996 | + | EXACTMASS 448.17333249399996 |
| − | AVERAGEMASS 448.46302000000003 | + | AVERAGEMASS 448.46302000000003 |
| − | SMILES COc(c1C(=O)C=Cc(c2)c(OC)c(c(c2OC)OC)OC)c(c(cc(OC)1)OC)OC | + | SMILES COc(c1C(=O)C=Cc(c2)c(OC)c(c(c2OC)OC)OC)c(c(cc(OC)1)OC)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 33 0 0 0 0 0 0 0 0999 V2000
-2.6887 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6887 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1324 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5761 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5761 0.3185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1324 0.6396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0198 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4635 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0926 -0.6450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6487 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2097 -0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7707 -0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7707 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2097 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6487 0.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0198 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6367 -0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5027 -1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5689 -0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8544 -1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7101 0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1559 1.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6367 0.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5027 1.3239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7756 -0.8097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4901 -1.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0460 0.9372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5461 1.8032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8544 1.2127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4180 2.1124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4901 1.2222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9288 2.1208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
12 17 1 0 0 0 0
17 18 1 0 0 0 0
3 19 1 0 0 0 0
19 20 1 0 0 0 0
5 21 1 0 0 0 0
21 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
11 25 1 0 0 0 0
25 26 1 0 0 0 0
1 27 1 0 0 0 0
27 28 1 0 0 0 0
6 29 1 0 0 0 0
29 30 1 0 0 0 0
14 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 8 SUP
M SLB 1 8 8
M SAL 8 2 31 32
M SBL 8 1 32
M SMT 8 OCH3
M SVB 8 32 1.4901 1.2222
M STY 1 7 SUP
M SLB 1 7 7
M SAL 7 2 29 30
M SBL 7 1 30
M SMT 7 OCH3
M SVB 7 30 -1.8544 1.2127
M STY 1 6 SUP
M SLB 1 6 6
M SAL 6 2 27 28
M SBL 6 1 28
M SMT 6 OCH3
M SVB 6 28 -3.046 0.9372
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 25 26
M SBL 5 1 26
M SMT 5 OCH3
M SVB 5 26 0.7756 -0.8097
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 23 24
M SBL 4 1 24
M SMT 4 OCH3
M SVB 4 24 2.3315 0.6477
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 21 22
M SBL 3 1 22
M SMT 3 OCH3
M SVB 3 22 -1.02 0.6395
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 19 20
M SBL 2 1 20
M SMT 2 OCH3
M SVB 2 20 -2.5689 -0.8056
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 17 18
M SBL 1 1 18
M SMT 1 OCH3
M SVB 1 18 1.9743 -0.5301
S SKP 8
ID FL1CELNS0002
KNApSAcK_ID C00006992
NAME 2,3,4,5,2',3',4',6'-Octamethoxychalcone
CAS_RN 121697-04-9
FORMULA C23H28O9
EXACTMASS 448.17333249399996
AVERAGEMASS 448.46302000000003
SMILES COc(c1C(=O)C=Cc(c2)c(OC)c(c(c2OC)OC)OC)c(c(cc(OC)1)OC)OC
M END