Mol:FL1CBAGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 46 0 0 0 0 0 0 0 0999 V2000 | + | 43 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2366 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2366 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2366 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2366 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5221 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5221 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1924 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1924 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1924 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1924 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5221 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5221 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9069 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9069 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6213 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6213 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6213 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6213 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3358 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3358 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0503 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0503 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7647 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7647 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7647 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7647 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0503 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0503 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3358 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3358 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9069 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9069 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0188 0.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0188 0.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5301 0.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5301 0.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7596 0.4309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7596 0.4309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9460 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9460 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4347 0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4347 0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2052 0.6621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2052 0.6621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4257 1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4257 1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4460 0.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4460 0.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0046 0.3433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0046 0.3433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8597 -0.2241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8597 -0.2241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5221 -2.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5221 -2.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9233 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9233 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0139 1.5323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0139 1.5323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4460 0.7585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4460 0.7585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8213 -0.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8213 -0.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8213 0.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8213 0.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2512 2.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2512 2.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7399 1.5377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7399 1.5377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5104 1.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5104 1.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3240 1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3240 1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8353 2.3602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8353 2.3602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0648 2.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0648 2.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8443 2.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8443 2.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8240 1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8240 1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2654 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2654 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4103 1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4103 1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2561 2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2561 2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 17 24 1 0 0 0 0 | + | 17 24 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 3 27 1 0 0 0 0 | + | 3 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 29 23 1 0 0 0 0 | + | 29 23 1 0 0 0 0 |
| − | 13 30 1 0 0 0 0 | + | 13 30 1 0 0 0 0 |
| − | 5 31 1 0 0 0 0 | + | 5 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 28 20 1 0 0 0 0 | + | 28 20 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 43 39 1 0 0 0 0 | + | 43 39 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CBAGS0004 | + | ID FL1CBAGS0004 |
| − | KNApSAcK_ID C00014506 | + | KNApSAcK_ID C00014506 |
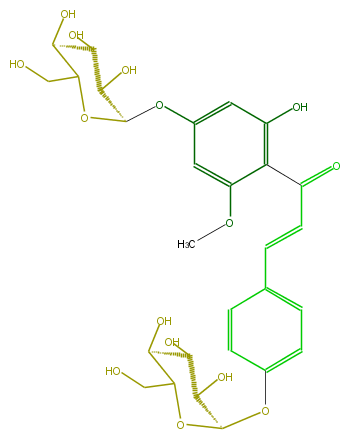
| − | NAME Helichrysetin 4,4'-di-O-alpha-glucoside;4,2',4'-Trihydroxy-6'-methoxychalcone 4,4'-di-O-alpha-glucoside | + | NAME Helichrysetin 4,4'-di-O-alpha-glucoside;4,2',4'-Trihydroxy-6'-methoxychalcone 4,4'-di-O-alpha-glucoside |
| − | CAS_RN 203193-16-2 | + | CAS_RN 203193-16-2 |
| − | FORMULA C28H34O15 | + | FORMULA C28H34O15 |
| − | EXACTMASS 610.189770418 | + | EXACTMASS 610.189770418 |
| − | AVERAGEMASS 610.56056 | + | AVERAGEMASS 610.56056 |
| − | SMILES O(C(Oc(c2)cc(OC)c(C(=O)C=Cc(c3)ccc(OC(O4)C(O)C(C(O)C4CO)O)c3)c(O)2)1)C(CO)C(O)C(C1O)O | + | SMILES O(C(Oc(c2)cc(OC)c(C(=O)C=Cc(c3)ccc(OC(O4)C(O)C(C(O)C4CO)O)c3)c(O)2)1)C(CO)C(O)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 46 0 0 0 0 0 0 0 0999 V2000
-1.2366 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2366 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5221 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1924 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1924 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5221 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9069 -2.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6213 -1.6973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6213 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3358 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0503 -0.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7647 -0.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7647 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0503 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3358 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9069 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0188 0.8002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5301 0.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7596 0.4309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9460 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4347 0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2052 0.6621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4257 1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4460 0.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0046 0.3433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8597 -0.2241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5221 -2.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9233 -0.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0139 1.5323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4460 0.7585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8213 -0.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8213 0.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2512 2.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7399 1.5377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5104 1.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3240 1.6951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8353 2.3602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0648 2.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8443 2.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8240 1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2654 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4103 1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2561 2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 18 1 1 0 0 0
18 19 1 1 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 17 1 0 0 0 0
22 23 1 0 0 0 0
17 24 1 0 0 0 0
18 25 1 0 0 0 0
19 26 1 0 0 0 0
3 27 1 0 0 0 0
1 28 1 0 0 0 0
29 23 1 0 0 0 0
13 30 1 0 0 0 0
5 31 1 0 0 0 0
31 32 1 0 0 0 0
28 20 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
43 39 1 0 0 0 0
30 36 1 0 0 0 0
S SKP 8
ID FL1CBAGS0004
KNApSAcK_ID C00014506
NAME Helichrysetin 4,4'-di-O-alpha-glucoside;4,2',4'-Trihydroxy-6'-methoxychalcone 4,4'-di-O-alpha-glucoside
CAS_RN 203193-16-2
FORMULA C28H34O15
EXACTMASS 610.189770418
AVERAGEMASS 610.56056
SMILES O(C(Oc(c2)cc(OC)c(C(=O)C=Cc(c3)ccc(OC(O4)C(O)C(C(O)C4CO)O)c3)c(O)2)1)C(CO)C(O)C(C1O)O
M END