Mol:FL1C3AGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 45 0 0 0 0 0 0 0 0999 V2000 | + | 42 45 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3472 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3472 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3472 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3472 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0617 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0617 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7762 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7762 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7762 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7762 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0617 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0617 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4906 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4906 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2051 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2051 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2051 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2051 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9196 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9196 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6341 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6341 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3485 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3485 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3485 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3485 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9196 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9196 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4906 -2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4906 -2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0371 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0371 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9762 0.2975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9762 0.2975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4983 -0.3753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4983 -0.3753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7231 -0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7231 -0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0882 -0.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0882 -0.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3896 0.4293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3896 0.4293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1649 0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1649 0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3761 0.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3761 0.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8799 0.9455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8799 0.9455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4119 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4119 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9693 -0.1595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9693 -0.1595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8338 -0.7454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8338 -0.7454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0617 -2.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0617 -2.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6605 0.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6605 0.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6473 -1.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6473 -1.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4943 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4943 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0728 2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0728 2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3181 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3181 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7396 0.7526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7396 0.7526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5634 0.7637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5634 0.7637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9662 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9662 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7912 1.4948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7912 1.4948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2133 0.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2133 0.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8104 0.0659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8104 0.0659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9855 0.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9855 0.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0371 0.7970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0371 0.7970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 21 20 1 1 0 0 0 | + | 21 20 1 1 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 2 31 1 0 0 0 0 | + | 2 31 1 0 0 0 0 |
| − | 30 21 1 0 0 0 0 | + | 30 21 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3AGS0002 | + | ID FL1C3AGS0002 |
| − | KNApSAcK_ID C00014500 | + | KNApSAcK_ID C00014500 |
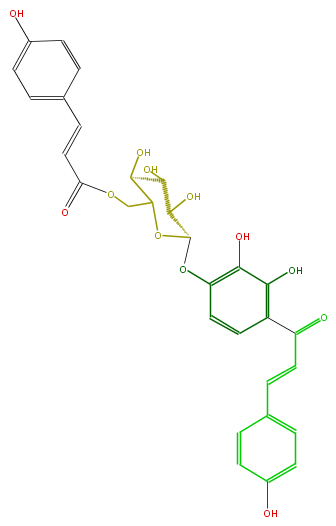
| − | NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(6''-O-p-coumaroyl)glucoside | + | NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(6''-O-p-coumaroyl)glucoside |
| − | CAS_RN 186145-46-0 | + | CAS_RN 186145-46-0 |
| − | FORMULA C30H28O12 | + | FORMULA C30H28O12 |
| − | EXACTMASS 580.15807636 | + | EXACTMASS 580.15807636 |
| − | AVERAGEMASS 580.53612 | + | AVERAGEMASS 580.53612 |
| − | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c1)c(O)c(c(C(C=Cc(c2)ccc(c2)O)=O)c1)O)O | + | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c1)c(O)c(c(C(C=Cc(c2)ccc(c2)O)=O)c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 45 0 0 0 0 0 0 0 0999 V2000
1.3472 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3472 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0617 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7762 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7762 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0617 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4906 -1.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2051 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2051 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9196 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6341 -0.0948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3485 0.3177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3485 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9196 1.1427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4906 -2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.0371 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9762 0.2975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4983 -0.3753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7231 -0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0882 -0.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3896 0.4293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1649 0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3761 0.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8799 0.9455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4119 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9693 -0.1595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8338 -0.7454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0617 -2.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6605 0.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6473 -1.3239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4943 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0728 2.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3181 1.4605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7396 0.7526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5634 0.7637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9662 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7912 1.4948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2133 0.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8104 0.0659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9855 0.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.0371 0.7970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
13 17 1 0 0 0 0
18 19 1 1 0 0 0
19 20 1 1 0 0 0
21 20 1 1 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
18 26 1 0 0 0 0
19 27 1 0 0 0 0
20 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
2 31 1 0 0 0 0
30 21 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
25 32 1 0 0 0 0
S SKP 8
ID FL1C3AGS0002
KNApSAcK_ID C00014500
NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(6''-O-p-coumaroyl)glucoside
CAS_RN 186145-46-0
FORMULA C30H28O12
EXACTMASS 580.15807636
AVERAGEMASS 580.53612
SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c1)c(O)c(c(C(C=Cc(c2)ccc(c2)O)=O)c1)O)O
M END