Mol:FL1C3AGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 45 0 0 0 0 0 0 0 0999 V2000 | + | 42 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9654 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9654 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9654 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9654 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2509 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2509 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4636 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4636 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4636 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4636 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2509 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2509 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1781 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1781 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8925 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8925 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8925 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8925 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6070 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6070 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3215 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3215 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0359 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0359 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0359 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0359 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3215 3.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3215 3.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6070 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6070 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1781 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1781 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7245 3.6825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7245 3.6825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2887 2.4397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2887 2.4397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8109 1.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8109 1.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0357 2.0500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0357 2.0500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2244 1.8986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2244 1.8986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7022 2.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7022 2.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4775 2.2884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4775 2.2884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6887 2.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6887 2.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1925 3.0877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1925 3.0877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7245 1.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7245 1.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2819 1.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2819 1.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1464 1.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1464 1.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2509 0.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2509 0.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6521 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6521 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6653 0.8183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6653 0.8183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9910 0.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9910 0.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7109 1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7109 1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9782 0.0007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9782 0.0007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2583 -0.4001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2583 -0.4001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2454 -1.2238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2454 -1.2238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9534 -1.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9534 -1.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9405 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9405 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2197 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2197 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5117 -2.4500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5117 -2.4500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5246 -1.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5246 -1.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2068 -3.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2068 -3.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 21 20 1 1 0 0 0 | + | 21 20 1 1 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 2 31 1 0 0 0 0 | + | 2 31 1 0 0 0 0 |
| − | 30 21 1 0 0 0 0 | + | 30 21 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3AGS0001 | + | ID FL1C3AGS0001 |
| − | KNApSAcK_ID C00014499 | + | KNApSAcK_ID C00014499 |
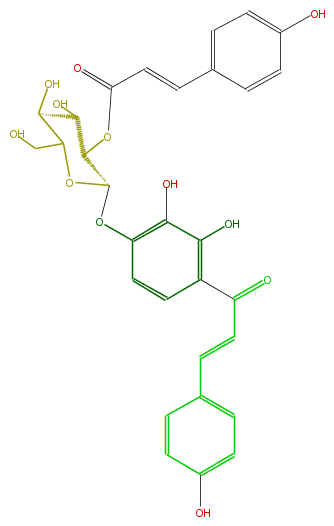
| − | NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(2''-O-p-coumaroyl)glucoside | + | NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(2''-O-p-coumaroyl)glucoside |
| − | CAS_RN 597550-50-0 | + | CAS_RN 597550-50-0 |
| − | FORMULA C30H28O12 | + | FORMULA C30H28O12 |
| − | EXACTMASS 580.15807636 | + | EXACTMASS 580.15807636 |
| − | AVERAGEMASS 580.53612 | + | AVERAGEMASS 580.53612 |
| − | SMILES c(C=CC(c(c2O)ccc(OC(C(OC(C=Cc(c4)ccc(c4)O)=O)3)OC(CO)C(O)C(O)3)c2O)=O)(c1)ccc(c1)O | + | SMILES c(C=CC(c(c2O)ccc(OC(C(OC(C=Cc(c4)ccc(c4)O)=O)3)OC(CO)C(O)C(O)3)c2O)=O)(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 45 0 0 0 0 0 0 0 0999 V2000
-0.9654 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9654 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2509 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4636 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4636 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2509 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1781 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8925 1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8925 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6070 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3215 2.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0359 2.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0359 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3215 3.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6070 3.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1781 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7245 3.6825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2887 2.4397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8109 1.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0357 2.0500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2244 1.8986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7022 2.5715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4775 2.2884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6887 2.8569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1925 3.0877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7245 1.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2819 1.9827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1464 1.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2509 0.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6521 2.4439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6653 0.8183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9910 0.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7109 1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9782 0.0007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2583 -0.4001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2454 -1.2238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9534 -1.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9405 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2197 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5117 -2.4500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5246 -1.6251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2068 -3.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
13 17 1 0 0 0 0
18 19 1 1 0 0 0
19 20 1 1 0 0 0
21 20 1 1 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
18 26 1 0 0 0 0
19 27 1 0 0 0 0
20 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
2 31 1 0 0 0 0
30 21 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
28 32 1 0 0 0 0
S SKP 8
ID FL1C3AGS0001
KNApSAcK_ID C00014499
NAME 4,2',3',4'-Tetrahydroxychalcone 4'-O-(2''-O-p-coumaroyl)glucoside
CAS_RN 597550-50-0
FORMULA C30H28O12
EXACTMASS 580.15807636
AVERAGEMASS 580.53612
SMILES c(C=CC(c(c2O)ccc(OC(C(OC(C=Cc(c4)ccc(c4)O)=O)3)OC(CO)C(O)C(O)3)c2O)=O)(c1)ccc(c1)O
M END