Mol:FL1AUNGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0354 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0354 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0354 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0354 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5546 1.7349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5546 1.7349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0738 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0738 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0738 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0738 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5546 0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5546 0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5348 0.6503 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5348 0.6503 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8872 1.1353 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.8872 1.1353 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.5348 1.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5348 1.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5930 1.7349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5930 1.7349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4745 1.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4745 1.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7673 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7673 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4027 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4027 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7204 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7204 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4027 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4027 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7673 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7673 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4496 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4496 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7204 3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7204 3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7241 -0.0314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7241 -0.0314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7741 -0.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7741 -0.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0418 -0.4663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0418 -0.4663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5270 -0.6134 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5270 -0.6134 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0092 -0.4663 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.0092 -0.4663 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.2586 -0.9299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2586 -0.9299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2562 -0.7828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.2562 -0.7828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.9717 -0.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9717 -0.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2643 -0.2746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2643 -0.2746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5916 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5916 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2130 -1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2130 -1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1134 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1134 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1200 -2.5688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1200 -2.5688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7652 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7652 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0911 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0911 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7428 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7428 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0245 -2.2409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0245 -2.2409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0687 -3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0687 -3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7204 -3.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7204 -3.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0418 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0418 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0418 -0.4663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0418 -0.4663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 2 1 0 0 0 0 | + | 9 2 1 0 0 0 0 |
| − | 4 10 1 0 0 0 0 | + | 4 10 1 0 0 0 0 |
| − | 8 11 1 6 0 0 0 | + | 8 11 1 6 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 21 20 1 1 0 0 0 | + | 21 20 1 1 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 20 1 1 0 0 0 | + | 25 20 1 1 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 19 22 1 0 0 0 0 | + | 19 22 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 7 19 1 1 0 0 0 | + | 7 19 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 41 1.1706 -0.6725 | + | M SVB 2 41 1.1706 -0.6725 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 39 -2.0687 -3.2933 | + | M SVB 1 39 -2.0687 -3.2933 |
| − | S SKP 8 | + | S SKP 8 |
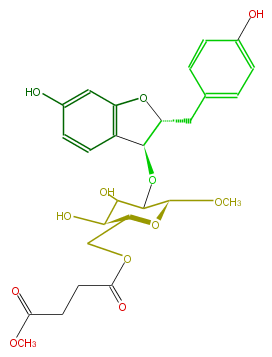
| − | ID FL1AUNGS0002 | + | ID FL1AUNGS0002 |
| − | KNApSAcK_ID C00008063 | + | KNApSAcK_ID C00008063 |
| − | NAME Ambofuracin | + | NAME Ambofuracin |
| − | CAS_RN 82358-50-7 | + | CAS_RN 82358-50-7 |
| − | FORMULA C27H32O12 | + | FORMULA C27H32O12 |
| − | EXACTMASS 548.189376488 | + | EXACTMASS 548.189376488 |
| − | AVERAGEMASS 548.5357799999999 | + | AVERAGEMASS 548.5357799999999 |
| − | SMILES c(c34)(ccc(c4)O)[C@@H]([C@H](O3)Cc(c2)ccc(O)c2)OC([C@H]1OC)C(O)[C@@H]([C@H](COC(CCC(=O)OC)=O)O1)O | + | SMILES c(c34)(ccc(c4)O)[C@@H]([C@H](O3)Cc(c2)ccc(O)c2)OC([C@H]1OC)C(O)[C@@H]([C@H](COC(CCC(=O)OC)=O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-0.0354 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0354 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5546 1.7349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0738 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0738 0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5546 0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5348 0.6503 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8872 1.1353 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.5348 1.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5930 1.7349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4745 1.1353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7673 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4027 1.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7204 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4027 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7673 2.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4496 2.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7204 3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7241 -0.0314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7741 -0.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0418 -0.4663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5270 -0.6134 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0092 -0.4663 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.2586 -0.9299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2562 -0.7828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.9717 -0.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2643 -0.2746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5916 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2130 -1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1134 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1200 -2.5688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7652 -2.1644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0911 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7428 -2.7289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0245 -2.2409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0687 -3.2933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7204 -3.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0418 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0418 -0.4663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
1 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 2 1 0 0 0 0
4 10 1 0 0 0 0
8 11 1 6 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
21 20 1 1 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 20 1 1 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
25 28 1 0 0 0 0
19 22 1 0 0 0 0
28 29 1 0 0 0 0
7 19 1 1 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
36 37 1 0 0 0 0
21 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 41
M SMT 2 OCH3
M SVB 2 41 1.1706 -0.6725
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 39
M SMT 1 OCH3
M SVB 1 39 -2.0687 -3.2933
S SKP 8
ID FL1AUNGS0002
KNApSAcK_ID C00008063
NAME Ambofuracin
CAS_RN 82358-50-7
FORMULA C27H32O12
EXACTMASS 548.189376488
AVERAGEMASS 548.5357799999999
SMILES c(c34)(ccc(c4)O)[C@@H]([C@H](O3)Cc(c2)ccc(O)c2)OC([C@H]1OC)C(O)[C@@H]([C@H](COC(CCC(=O)OC)=O)O1)O
M END