Mol:FL1AAGGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5792 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5792 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5792 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5792 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0984 1.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0984 1.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6177 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6177 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6177 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6177 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0984 0.0181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0984 0.0181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0090 0.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0090 0.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3434 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3434 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0090 1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0090 1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1368 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1368 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7851 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7851 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6690 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6690 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9693 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9693 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5699 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5699 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8701 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8701 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5699 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5699 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9693 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9693 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4696 0.6177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4696 0.6177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1353 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1353 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8696 1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8696 1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0984 -0.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0984 -0.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8696 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8696 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9558 -0.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9558 -0.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5846 -1.3507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5846 -1.3507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0501 -1.1428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0501 -1.1428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5344 -1.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5344 -1.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9091 -0.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9091 -0.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3768 -1.0092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3768 -1.0092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4696 -1.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4696 -1.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1633 -1.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1633 -1.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7439 -1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7439 -1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7752 -0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7752 -0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9783 -0.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9783 -0.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 2 1 0 0 0 0 | + | 9 2 1 0 0 0 0 |
| − | 4 10 1 0 0 0 0 | + | 4 10 1 0 0 0 0 |
| − | 8 11 2 0 0 0 0 | + | 8 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 7 19 2 0 0 0 0 | + | 7 19 2 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 35 -7.5529 6.7739 | + | M SBV 1 35 -7.5529 6.7739 |
| − | S SKP 8 | + | S SKP 8 |
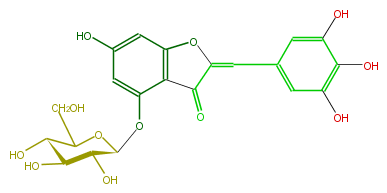
| − | ID FL1AAGGS0001 | + | ID FL1AAGGS0001 |
| − | KNApSAcK_ID C00008059 | + | KNApSAcK_ID C00008059 |
| − | NAME Bractein | + | NAME Bractein |
| − | CAS_RN 22684-08-8 | + | CAS_RN 22684-08-8 |
| − | FORMULA C21H20O12 | + | FORMULA C21H20O12 |
| − | EXACTMASS 464.095476104 | + | EXACTMASS 464.095476104 |
| − | AVERAGEMASS 464.37629999999996 | + | AVERAGEMASS 464.37629999999996 |
| − | SMILES C(C1Oc(c42)cc(O)cc2oc(c4=O)=Cc(c3)cc(O)c(O)c3O)(O)C(O)C(O)C(O1)CO | + | SMILES C(C1Oc(c42)cc(O)cc2oc(c4=O)=Cc(c3)cc(O)c(O)c3O)(O)C(O)C(O)C(O1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.5792 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5792 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0984 1.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6177 0.9174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6177 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0984 0.0181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0090 0.1326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3434 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0090 1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1368 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7851 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6690 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9693 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5699 0.0975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8701 0.6177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5699 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9693 1.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4696 0.6177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1353 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8696 1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0984 -0.5814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8696 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9558 -0.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5846 -1.3507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0501 -1.1428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5344 -1.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9091 -0.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3768 -1.0092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4696 -1.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1633 -1.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7439 -1.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7752 -0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9783 -0.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
1 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 2 1 0 0 0 0
4 10 1 0 0 0 0
8 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
7 19 2 0 0 0 0
16 20 1 0 0 0 0
6 21 1 0 0 0 0
14 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 21 1 0 0 0 0
28 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 ^CH2OH
M SBV 1 35 -7.5529 6.7739
S SKP 8
ID FL1AAGGS0001
KNApSAcK_ID C00008059
NAME Bractein
CAS_RN 22684-08-8
FORMULA C21H20O12
EXACTMASS 464.095476104
AVERAGEMASS 464.37629999999996
SMILES C(C1Oc(c42)cc(O)cc2oc(c4=O)=Cc(c3)cc(O)c(O)c3O)(O)C(O)C(O)C(O1)CO
M END