Mol:FL1A3CGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 44 0 0 0 0 0 0 0 0999 V2000 | + | 41 44 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9753 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9753 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9753 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9753 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2608 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2608 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2608 1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2608 1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4536 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4536 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4536 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4536 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1679 1.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1679 1.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8842 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8842 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8833 0.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8833 0.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6597 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6597 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4237 0.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4237 0.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4347 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4347 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1548 -0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1548 -0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8635 0.1519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8635 0.1519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8522 0.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8522 0.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1322 1.3793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1322 1.3793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1486 -0.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1486 -0.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6610 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6610 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8900 -0.8068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8900 -0.8068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0766 -0.9465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0766 -0.9465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5641 -0.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5641 -0.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3352 -0.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3352 -0.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5547 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5547 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0617 0.2141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0617 0.2141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5766 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5766 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1351 -0.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1351 -0.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9912 -1.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9912 -1.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2608 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2608 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5131 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5131 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1679 1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1679 1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1548 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1548 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0617 0.7283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0617 0.7283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4936 0.9777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4936 0.9777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5898 1.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5898 1.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5852 -0.2838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5852 -0.2838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1351 -1.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1351 -1.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7627 -1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7627 -1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7821 -1.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7821 -1.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2603 -0.9680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2603 -0.9680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4015 -0.4411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4015 -0.4411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5131 -1.4058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5131 -1.4058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 5 2 0 0 0 0 | + | 3 5 2 0 0 0 0 |
| − | 6 4 2 0 0 0 0 | + | 6 4 2 0 0 0 0 |
| − | 4 1 1 0 0 0 0 | + | 4 1 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 5 9 1 0 0 0 0 | + | 5 9 1 0 0 0 0 |
| − | 8 10 2 0 0 0 0 | + | 8 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 17 25 1 0 0 0 0 | + | 17 25 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 14 29 1 0 0 0 0 | + | 14 29 1 0 0 0 0 |
| − | 7 30 2 0 0 0 0 | + | 7 30 2 0 0 0 0 |
| − | 13 31 1 0 0 0 0 | + | 13 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 2 35 1 0 0 0 0 | + | 2 35 1 0 0 0 0 |
| − | 35 20 1 0 0 0 0 | + | 35 20 1 0 0 0 0 |
| − | 26 36 1 0 0 0 0 | + | 26 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1A3CGS0011 | + | ID FL1A3CGS0011 |
| − | KNApSAcK_ID C00014667 | + | KNApSAcK_ID C00014667 |
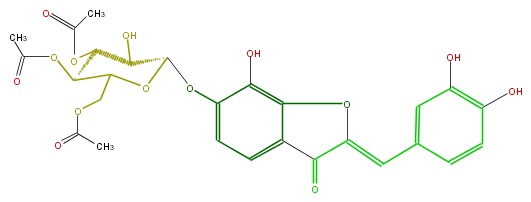
| − | NAME 6,7,3',4'-Tetrahydroxyaurone 6-O-(3'',4'',6''-tri-O-acetylglucoside);Maritimetin 6-O-(3'',4'',6''-tri-O-acetylglucoside) | + | NAME 6,7,3',4'-Tetrahydroxyaurone 6-O-(3'',4'',6''-tri-O-acetylglucoside);Maritimetin 6-O-(3'',4'',6''-tri-O-acetylglucoside) |
| − | CAS_RN 194600-18-5 | + | CAS_RN 194600-18-5 |
| − | FORMULA C27H26O14 | + | FORMULA C27H26O14 |
| − | EXACTMASS 574.13225554 | + | EXACTMASS 574.13225554 |
| − | AVERAGEMASS 574.48694 | + | AVERAGEMASS 574.48694 |
| − | SMILES c(c23)cc(c(O)c2oc(=Cc(c4)cc(c(O)c4)O)c3=O)OC(O1)C(O)C(C(C1COC(C)=O)OC(C)=O)OC(C)=O | + | SMILES c(c23)cc(c(O)c2oc(=Cc(c4)cc(c(O)c4)O)c3=O)OC(O1)C(O)C(C(C1COC(C)=O)OC(C)=O)OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 44 0 0 0 0 0 0 0 0999 V2000
-0.9753 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9753 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2608 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2608 1.3058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4536 0.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4536 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1679 1.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8842 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8833 0.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6597 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4237 0.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4347 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1548 -0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8635 0.1519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8522 0.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1322 1.3793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1486 -0.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6610 -1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8900 -0.8068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0766 -0.9465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5641 -0.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3352 -0.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5547 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0617 0.2141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5766 -0.9680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1351 -0.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9912 -1.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2608 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5131 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1679 1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1548 -0.9723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0617 0.7283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4936 0.9777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5898 1.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5852 -0.2838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1351 -1.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7627 -1.9493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7821 -1.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2603 -0.9680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4015 -0.4411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5131 -1.4058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 5 2 0 0 0 0
6 4 2 0 0 0 0
4 1 1 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
5 9 1 0 0 0 0
8 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
17 18 1 1 0 0 0
18 19 1 1 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 17 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
17 25 1 0 0 0 0
18 26 1 0 0 0 0
19 27 1 0 0 0 0
3 28 1 0 0 0 0
14 29 1 0 0 0 0
7 30 2 0 0 0 0
13 31 1 0 0 0 0
24 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
2 35 1 0 0 0 0
35 20 1 0 0 0 0
26 36 1 0 0 0 0
36 37 2 0 0 0 0
36 38 1 0 0 0 0
25 39 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
S SKP 8
ID FL1A3CGS0011
KNApSAcK_ID C00014667
NAME 6,7,3',4'-Tetrahydroxyaurone 6-O-(3'',4'',6''-tri-O-acetylglucoside);Maritimetin 6-O-(3'',4'',6''-tri-O-acetylglucoside)
CAS_RN 194600-18-5
FORMULA C27H26O14
EXACTMASS 574.13225554
AVERAGEMASS 574.48694
SMILES c(c23)cc(c(O)c2oc(=Cc(c4)cc(c(O)c4)O)c3=O)OC(O1)C(O)C(C(C1COC(C)=O)OC(C)=O)OC(C)=O
M END