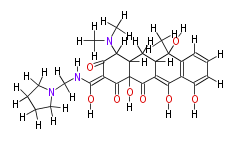
Mol:COX00079
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 6420073 | + | 6420073 |
| − | CDK 9/16/09,17:12 | + | CDK 9/16/09,17:12 |
| − | + | ||
| − | 71 75 0 0 0 0 0 0 0 0999 V2000 | + | 71 75 0 0 0 0 0 0 0 0999 V2000 |
| − | 8.8481 1.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8481 1.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9640 -1.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9640 -1.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7141 2.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7141 2.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4642 1.9818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4642 1.9818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4625 2.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4625 2.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1840 -0.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1840 -0.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.2997 2.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.2997 2.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1878 2.0995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1878 2.0995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9657 -1.9592 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9657 -1.9592 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3160 0.6029 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3160 0.6029 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5840 0.6095 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5840 0.6095 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8481 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8481 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5801 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5801 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7141 -0.9246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7141 -0.9246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8481 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8481 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9541 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9541 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4740 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4740 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5801 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5801 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7141 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7141 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9541 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9541 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3801 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.3801 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0480 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0480 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4740 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4740 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0480 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0480 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3801 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.3801 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9641 -1.8195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9641 -1.8195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3113 -1.0024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3113 -1.0024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1055 -2.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1055 -2.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8374 -2.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8374 -2.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3113 1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3113 1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1840 1.0995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1840 1.0995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2550 -0.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2550 -0.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2550 0.6179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2550 0.6179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4519 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4519 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6720 1.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6720 1.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4756 -0.3846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4756 -0.3846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4967 -0.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4967 -0.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8546 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8546 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5735 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5735 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3155 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3155 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1126 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1126 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4952 -1.2620 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4952 -1.2620 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.4307 -1.5033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.4307 -1.5033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6479 -2.3528 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6479 -2.3528 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4974 -2.1357 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4974 -2.1357 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5381 2.1123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5381 2.1123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6478 -2.3644 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6478 -2.3644 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3041 -1.6223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3041 -1.6223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7893 -1.9359 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7893 -1.9359 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5722 -2.7854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5722 -2.7854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4217 -3.0025 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4217 -3.0025 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5337 -2.9897 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5337 -2.9897 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3779 -2.7530 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3779 -2.7530 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1412 -1.9087 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1412 -1.9087 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9958 2.4261 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9958 2.4261 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7908 -0.7792 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7908 -0.7792 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7908 0.9299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7908 0.9299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3136 -0.0171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3136 -0.0171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8523 1.5796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8523 1.5796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0552 1.5827 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0552 1.5827 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9841 1.5555 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9841 1.5555 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1718 1.3861 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1718 1.3861 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5380 -1.0014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5380 -1.0014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0956 -0.3870 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0956 -0.3870 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5869 0.7416 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5869 0.7416 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4970 -0.0833 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4970 -0.0833 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9293 -0.8387 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9293 -0.8387 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6860 -1.1791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6860 -1.1791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6521 2.4116 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6521 2.4116 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8330 2.4692 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8330 2.4692 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
15 1 1 6 0 0 0 | 15 1 1 6 0 0 0 | ||
1 47 1 0 0 0 0 | 1 47 1 0 0 0 0 | ||
| Line 149: | Line 149: | ||
38 68 1 0 0 0 0 | 38 68 1 0 0 0 0 | ||
38 69 1 0 0 0 0 | 38 69 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
| − | NAME Rolitetracycline | + | NAME Rolitetracycline |
| + | ID COX00079 | ||
| + | FORMULA C27H33N3O8 | ||
| + | EXACTMASS 527.226765047 | ||
| + | AVERAGEMASS 527.56634 | ||
| + | SMILES [H]C([H])(N(C5([H])[H])C(C(C([H])([H])5)([H])[H])([H])[H])N(C(O[H])=C(C(=O)1)C(C(C(=O)4)(C(C(C(C4=3)(C(C([H])([H])[H])(O[H])c(c(C(O[H])3)2)c(c(c(c(O[H])2)[H])[H])[H])[H])([H])[H])(C(N(C([H])([H])[H])C([H])([H])[H])1[H])[H])O[H])=O)[H] | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
6420073
CDK 9/16/09,17:12
71 75 0 0 0 0 0 0 0 0999 V2000
8.8481 1.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.9640 -1.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.7141 2.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.4642 1.9818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.4625 2.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1840 -0.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.2997 2.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1878 2.0995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.9657 -1.9592 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.3160 0.6029 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.5840 0.6095 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.8481 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5801 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7141 -0.9246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8481 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9541 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.4740 -0.9593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5801 0.5754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7141 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9541 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.3801 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0480 -0.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.4740 1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0480 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.3801 0.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.9641 -1.8195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3113 -1.0024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1055 -2.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8374 -2.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3113 1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1840 1.0995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2550 -0.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2550 0.6179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4519 1.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6720 1.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4756 -0.3846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4967 -0.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8546 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.5735 -1.2746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3155 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.1126 -1.3996 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4952 -1.2620 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.4307 -1.5033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.6479 -2.3528 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.4974 -2.1357 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.5381 2.1123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.6478 -2.3644 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.3041 -1.6223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7893 -1.9359 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5722 -2.7854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4217 -3.0025 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.5337 -2.9897 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3779 -2.7530 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.1412 -1.9087 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.9958 2.4261 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
14.7908 -0.7792 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
14.7908 0.9299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3136 -0.0171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.8523 1.5796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0552 1.5827 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9841 1.5555 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1718 1.3861 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5380 -1.0014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0956 -0.3870 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.5869 0.7416 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4970 -0.0833 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.9293 -0.8387 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.6860 -1.1791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6521 2.4116 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.8330 2.4692 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
15 1 1 6 0 0 0
1 47 1 0 0 0 0
17 2 1 1 0 0 0
2 48 1 0 0 0 0
3 19 2 0 0 0 0
4 20 2 0 0 0 0
5 23 1 0 0 0 0
5 56 1 0 0 0 0
6 22 2 0 0 0 0
7 30 1 0 0 0 0
7 71 1 0 0 0 0
8 31 1 0 0 0 0
8 70 1 0 0 0 0
16 9 1 6 0 0 0
9 28 1 0 0 0 0
9 29 1 0 0 0 0
10 31 1 0 0 0 0
10 34 1 0 0 0 0
10 59 1 0 0 0 0
11 34 1 0 0 0 0
11 35 1 0 0 0 0
11 36 1 0 0 0 0
12 14 1 0 0 0 0
12 15 1 0 0 0 0
12 16 1 0 0 0 0
12 39 1 0 0 0 0
13 14 1 0 0 0 0
13 17 1 0 0 0 0
13 18 1 0 0 0 0
13 40 1 0 0 0 0
14 41 1 0 0 0 0
14 42 1 0 0 0 0
15 19 1 0 0 0 0
15 20 1 0 0 0 0
16 22 1 0 0 0 0
16 43 1 0 0 0 0
17 21 1 0 0 0 0
17 26 1 0 0 0 0
18 19 1 0 0 0 0
18 23 2 0 0 0 0
20 24 1 0 0 0 0
21 25 2 0 0 0 0
21 27 1 0 0 0 0
22 24 1 0 0 0 0
23 25 1 0 0 0 0
24 31 2 0 0 0 0
25 30 1 0 0 0 0
26 44 1 0 0 0 0
26 45 1 0 0 0 0
26 46 1 0 0 0 0
27 32 2 0 0 0 0
27 49 1 0 0 0 0
28 50 1 0 0 0 0
28 51 1 0 0 0 0
28 52 1 0 0 0 0
29 53 1 0 0 0 0
29 54 1 0 0 0 0
29 55 1 0 0 0 0
30 33 2 0 0 0 0
32 33 1 0 0 0 0
32 57 1 0 0 0 0
33 58 1 0 0 0 0
34 60 1 0 0 0 0
34 61 1 0 0 0 0
35 37 1 0 0 0 0
35 62 1 0 0 0 0
35 63 1 0 0 0 0
36 38 1 0 0 0 0
36 64 1 0 0 0 0
36 65 1 0 0 0 0
37 38 1 0 0 0 0
37 66 1 0 0 0 0
37 67 1 0 0 0 0
38 68 1 0 0 0 0
38 69 1 0 0 0 0
S SKP 6
NAME Rolitetracycline
ID COX00079
FORMULA C27H33N3O8
EXACTMASS 527.226765047
AVERAGEMASS 527.56634
SMILES [H]C([H])(N(C5([H])[H])C(C(C([H])([H])5)([H])[H])([H])[H])N(C(O[H])=C(C(=O)1)C(C(C(=O)4)(C(C(C(C4=3)(C(C([H])([H])[H])(O[H])c(c(C(O[H])3)2)c(c(c(c(O[H])2)[H])[H])[H])[H])([H])[H])(C(N(C([H])([H])[H])C([H])([H])[H])1[H])[H])O[H])=O)[H]
M END