Mol:COX00077
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 60815 | + | 60815 |
| − | CDK 9/16/09,17:11 | + | CDK 9/16/09,17:11 |
| − | + | ||
| − | 55 56 0 0 0 0 0 0 0 0999 V2000 | + | 55 56 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7660 1.5616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7660 1.5616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3637 2.8433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3637 2.8433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1553 3.8777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1553 3.8777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7397 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7397 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4718 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4718 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4718 -0.7392 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4718 -0.7392 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9718 2.1268 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9718 2.1268 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3378 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3378 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3378 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3378 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7057 1.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7057 1.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4718 -1.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4718 -1.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7981 3.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7981 3.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 -2.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 -2.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5642 3.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5642 3.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6058 -3.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6058 -3.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3905 4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3905 4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7397 -4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7397 -4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3937 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3937 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9952 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9952 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9484 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9484 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5499 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5499 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9952 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9952 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3937 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3937 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5499 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5499 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9484 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9484 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0824 -1.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0824 -1.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6838 -2.3218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6838 -2.3218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9952 -2.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9952 -2.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3937 -1.6566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3937 -1.6566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1468 3.9665 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1468 3.9665 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8742 3.2175 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8742 3.2175 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1618 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1618 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1618 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1618 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0011 4.8469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0011 4.8469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2829 5.3498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2829 5.3498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7799 4.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7799 4.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3985 2.6793 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3985 2.6793 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5251 2.6029 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5251 2.6029 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6015 1.7294 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6015 1.7294 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7818 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7818 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7818 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7818 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5918 2.1268 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5918 2.1268 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1197 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1197 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7397 -5.3592 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7397 -5.3592 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3597 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3597 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 13 1 0 0 0 0 | 1 13 1 0 0 0 0 | ||
1 23 1 0 0 0 0 | 1 23 1 0 0 0 0 | ||
| Line 114: | Line 114: | ||
27 54 1 0 0 0 0 | 27 54 1 0 0 0 0 | ||
27 55 1 0 0 0 0 | 27 55 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
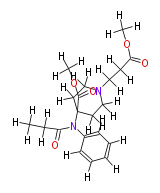
| − | NAME Remifentanil | + | NAME Remifentanil |
| + | ID COX00077 | ||
| + | FORMULA C20H28N2O5 | ||
| + | EXACTMASS 376.199822016 | ||
| + | AVERAGEMASS 376.44680000000005 | ||
| + | SMILES [H]C([H])([H])OC(=O)C([H])([H])C([H])([H])N(C([H])([H])1)C([H])([H])C([H])([H])C(C(=O)OC([H])([H])[H])(N(C(=O)C([H])([H])C([H])([H])[H])c(c([H])2)c([H])c([H])c([H])c([H])2)C([H])([H])1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
60815
CDK 9/16/09,17:11
55 56 0 0 0 0 0 0 0 0999 V2000
2.7660 1.5616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3637 2.8433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1553 3.8777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7397 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4718 -3.7392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4718 -0.7392 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.9718 2.1268 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3378 0.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3378 -0.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7057 1.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4718 -1.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7981 3.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 -2.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5642 3.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6058 -3.2392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3905 4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4718 2.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4718 1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9718 2.1268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7397 -4.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3937 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9952 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9484 0.6531 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5499 1.3434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9952 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.3937 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5499 -0.8218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9484 -0.1316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0824 -1.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.6838 -2.3218 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9952 -2.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.3937 -1.6566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1468 3.9665 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8742 3.2175 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1618 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1618 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0011 4.8469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.2829 5.3498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7799 4.6316 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3985 2.6793 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.5251 2.6029 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.6015 1.7294 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.7818 3.5298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.7818 0.7238 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.5918 2.1268 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1197 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.7397 -5.3592 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.3597 -4.7392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 13 1 0 0 0 0
1 23 1 0 0 0 0
2 13 2 0 0 0 0
3 16 2 0 0 0 0
4 21 1 0 0 0 0
4 27 1 0 0 0 0
5 21 2 0 0 0 0
6 11 1 0 0 0 0
6 12 1 0 0 0 0
6 14 1 0 0 0 0
7 8 1 0 0 0 0
7 15 1 0 0 0 0
7 16 1 0 0 0 0
8 9 1 0 0 0 0
8 10 1 0 0 0 0
8 13 1 0 0 0 0
9 11 1 0 0 0 0
9 28 1 0 0 0 0
9 29 1 0 0 0 0
10 12 1 0 0 0 0
10 30 1 0 0 0 0
10 31 1 0 0 0 0
11 32 1 0 0 0 0
11 33 1 0 0 0 0
12 34 1 0 0 0 0
12 35 1 0 0 0 0
14 17 1 0 0 0 0
14 36 1 0 0 0 0
14 37 1 0 0 0 0
15 19 2 0 0 0 0
15 20 1 0 0 0 0
16 18 1 0 0 0 0
17 21 1 0 0 0 0
17 38 1 0 0 0 0
17 39 1 0 0 0 0
18 22 1 0 0 0 0
18 40 1 0 0 0 0
18 41 1 0 0 0 0
19 24 1 0 0 0 0
19 42 1 0 0 0 0
20 25 2 0 0 0 0
20 43 1 0 0 0 0
22 44 1 0 0 0 0
22 45 1 0 0 0 0
22 46 1 0 0 0 0
23 47 1 0 0 0 0
23 48 1 0 0 0 0
23 49 1 0 0 0 0
24 26 2 0 0 0 0
24 50 1 0 0 0 0
25 26 1 0 0 0 0
25 51 1 0 0 0 0
26 52 1 0 0 0 0
27 53 1 0 0 0 0
27 54 1 0 0 0 0
27 55 1 0 0 0 0
S SKP 6
NAME Remifentanil
ID COX00077
FORMULA C20H28N2O5
EXACTMASS 376.199822016
AVERAGEMASS 376.44680000000005
SMILES [H]C([H])([H])OC(=O)C([H])([H])C([H])([H])N(C([H])([H])1)C([H])([H])C([H])([H])C(C(=O)OC([H])([H])[H])(N(C(=O)C([H])([H])C([H])([H])[H])c(c([H])2)c([H])c([H])c([H])c([H])2)C([H])([H])1
M END