Mol:COX00068
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 107807 | + | 107807 |
| − | CDK 9/16/09,17:9 | + | CDK 9/16/09,17:9 |
| − | + | ||
| − | 58 59 0 0 0 0 0 0 0 0999 V2000 | + | 58 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 6.6284 -1.3305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6284 -1.3305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4228 -4.1580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4228 -4.1580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4228 -2.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4228 -2.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2677 2.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2677 2.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9140 2.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9140 2.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3392 -2.4873 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3392 -2.4873 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2927 0.1581 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2927 0.1581 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3930 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3930 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3930 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3930 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3392 -4.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3392 -4.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5270 -4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5270 -4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5270 -2.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5270 -2.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6609 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6609 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6609 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6609 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6499 -1.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6499 -1.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9820 -0.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9820 -0.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0035 -0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0035 -0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6249 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6249 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6463 0.6961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6463 0.6961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9355 1.8529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9355 1.8529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9785 1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9785 1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 1.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 1.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5783 3.5477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5783 3.5477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9105 4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9105 4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4777 -4.5875 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4777 -4.5875 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4777 -1.9965 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4777 -1.9965 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8766 -4.4060 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8766 -4.4060 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0882 -4.6636 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0882 -4.6636 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2045 -2.7397 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2045 -2.7397 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1284 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1284 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9255 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9255 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1284 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1284 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9255 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9255 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4489 -4.3746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4489 -4.3746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0504 -3.6843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0504 -3.6843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4489 -2.2094 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4489 -2.2094 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0504 -2.8997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0504 -2.8997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5680 -0.3310 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5680 -0.3310 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8994 0.2859 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8994 0.2859 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1314 -1.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1314 -1.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3969 -1.1265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3969 -1.1265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8757 -0.3920 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8757 -0.3920 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0428 -4.1580 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0428 -4.1580 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2315 1.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2315 1.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8776 0.1209 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8776 0.1209 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1200 0.3685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1200 0.3685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7472 2.0157 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7472 2.0157 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5049 1.7681 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5049 1.7681 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1279 0.6276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1279 0.6276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3933 1.1064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3933 1.1064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8721 1.8409 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8721 1.8409 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9609 4.0356 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9609 4.0356 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1253 3.2557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1253 3.2557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4490 3.8779 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4490 3.8779 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4964 4.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4964 4.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3720 4.7061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3720 4.7061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 16 2 0 0 0 0 | 1 16 2 0 0 0 0 | ||
2 17 1 0 0 0 0 | 2 17 1 0 0 0 0 | ||
| Line 120: | Line 120: | ||
26 57 1 0 0 0 0 | 26 57 1 0 0 0 0 | ||
26 58 1 0 0 0 0 | 26 58 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
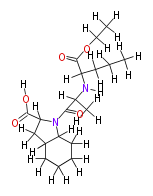
| − | NAME Perindopril | + | NAME Perindopril |
| + | ID COX00068 | ||
| + | FORMULA C19H32N2O5 | ||
| + | EXACTMASS 368.231122144 | ||
| + | AVERAGEMASS 368.46786000000003 | ||
| + | SMILES [H]OC(=O)C([H])(C([H])([H])1)N(C(=O)C([H])(C([H])([H])[H])N([H])C([H])(C(=O)OC([H])([H])C([H])([H])[H])C([H])([H])C([H])([H])C([H])([H])[H])C([H])(C([H])([H])2)C([H])(C([H])([H])C([H])([H])C([H])([H])2)1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
107807
CDK 9/16/09,17:9
58 59 0 0 0 0 0 0 0 0999 V2000
6.6284 -1.3305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.4228 -4.1580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.4228 -2.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2677 2.5972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9140 2.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3392 -2.4873 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.2927 0.1581 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.3930 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3930 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3392 -4.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5270 -4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5270 -2.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6609 -3.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6609 -2.7920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6499 -1.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9228 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9820 -0.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0035 -0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6249 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6463 0.6961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9355 1.8529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9785 1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 1.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5783 3.5477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9105 4.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4777 -4.5875 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4777 -1.9965 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8766 -4.4060 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0882 -4.6636 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.2045 -2.7397 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1284 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9255 -4.7670 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1284 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9255 -1.8171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.4489 -4.3746 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0504 -3.6843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.4489 -2.2094 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0504 -2.8997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5680 -0.3310 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8994 0.2859 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.1314 -1.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.3969 -1.1265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8757 -0.3920 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0428 -4.1580 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.2315 1.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8776 0.1209 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1200 0.3685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.7472 2.0157 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5049 1.7681 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1279 0.6276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3933 1.1064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.8721 1.8409 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9609 4.0356 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1253 3.2557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4490 3.8779 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4964 4.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3720 4.7061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 16 2 0 0 0 0
2 17 1 0 0 0 0
2 45 1 0 0 0 0
3 17 2 0 0 0 0
4 22 1 0 0 0 0
4 25 1 0 0 0 0
5 22 2 0 0 0 0
6 9 1 0 0 0 0
6 11 1 0 0 0 0
6 16 1 0 0 0 0
18 7 1 6 0 0 0
20 7 1 1 0 0 0
7 41 1 0 0 0 0
8 9 1 0 0 0 0
8 10 1 0 0 0 0
8 12 1 0 0 0 0
8 27 1 1 0 0 0
9 13 1 0 0 0 0
9 28 1 1 0 0 0
10 11 1 0 0 0 0
10 29 1 0 0 0 0
10 30 1 0 0 0 0
11 17 1 6 0 0 0
11 31 1 0 0 0 0
12 14 1 0 0 0 0
12 32 1 0 0 0 0
12 33 1 0 0 0 0
13 15 1 0 0 0 0
13 34 1 0 0 0 0
13 35 1 0 0 0 0
14 15 1 0 0 0 0
14 36 1 0 0 0 0
14 37 1 0 0 0 0
15 38 1 0 0 0 0
15 39 1 0 0 0 0
16 18 1 0 0 0 0
18 19 1 0 0 0 0
18 40 1 0 0 0 0
19 42 1 0 0 0 0
19 43 1 0 0 0 0
19 44 1 0 0 0 0
20 21 1 0 0 0 0
20 22 1 0 0 0 0
20 46 1 0 0 0 0
21 23 1 0 0 0 0
21 47 1 0 0 0 0
21 48 1 0 0 0 0
23 24 1 0 0 0 0
23 49 1 0 0 0 0
23 50 1 0 0 0 0
24 51 1 0 0 0 0
24 52 1 0 0 0 0
24 53 1 0 0 0 0
25 26 1 0 0 0 0
25 54 1 0 0 0 0
25 55 1 0 0 0 0
26 56 1 0 0 0 0
26 57 1 0 0 0 0
26 58 1 0 0 0 0
S SKP 6
NAME Perindopril
ID COX00068
FORMULA C19H32N2O5
EXACTMASS 368.231122144
AVERAGEMASS 368.46786000000003
SMILES [H]OC(=O)C([H])(C([H])([H])1)N(C(=O)C([H])(C([H])([H])[H])N([H])C([H])(C(=O)OC([H])([H])C([H])([H])[H])C([H])([H])C([H])([H])C([H])([H])[H])C([H])(C([H])([H])2)C([H])(C([H])([H])C([H])([H])C([H])([H])2)1
M END