Mol:COX00062
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4544 | + | 4544 |
| − | CDK 9/16/09,17:8 | + | CDK 9/16/09,17:8 |
| − | + | ||
| − | 53 57 0 0 0 0 0 0 0 0999 V2000 | + | 53 57 0 0 0 0 0 0 0 0999 V2000 |
| − | 6.5678 0.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5678 0.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5047 -0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5047 -0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3170 -1.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3170 -1.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3170 -3.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3170 -3.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9425 2.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9425 2.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8739 1.8634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8739 1.8634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9808 2.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9808 2.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8387 -1.9744 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8387 -1.9744 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7448 -1.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7448 -1.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7563 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7563 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6387 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6387 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8387 -3.0161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8387 -3.0161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6387 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6387 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7448 -3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7448 -3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9584 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9584 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5047 -1.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5047 -1.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9746 -1.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9746 -1.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2800 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2800 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5047 -3.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5047 -3.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3708 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3708 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3708 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3708 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2740 1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2740 1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9776 -0.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9776 -0.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6208 1.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6208 1.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3184 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3184 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6400 1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6400 1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9006 -2.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9006 -2.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3708 0.0048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3708 0.0048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2833 3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2833 3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.1933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.1933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2109 -1.1454 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2109 -1.1454 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3053 -0.7487 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3053 -0.7487 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2279 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2279 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6297 -3.5998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6297 -3.5998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3511 -4.0089 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3511 -4.0089 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1494 -3.9997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1494 -3.9997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2867 -0.9354 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2867 -0.9354 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4389 -1.1590 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4389 -1.1590 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6625 -2.0068 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6625 -2.0068 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5047 -4.1152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5047 -4.1152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7782 -0.6497 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7782 -0.6497 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7103 0.5685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7103 0.5685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3615 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3615 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3615 -2.0805 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3615 -2.0805 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6808 -0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6808 -0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9077 0.3148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9077 0.3148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0608 0.5417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0608 0.5417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7495 3.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7495 3.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8746 3.9961 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8746 3.9961 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8171 3.1212 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8171 3.1212 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8792 2.8014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8792 2.8014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3919 2.0724 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3919 2.0724 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1208 1.5852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1208 1.5852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 10 1 0 0 0 0 | 1 10 1 0 0 0 0 | ||
1 22 1 0 0 0 0 | 1 22 1 0 0 0 0 | ||
| Line 113: | Line 113: | ||
30 52 1 0 0 0 0 | 30 52 1 0 0 0 0 | ||
30 53 1 0 0 0 0 | 30 53 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
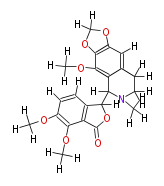
| − | NAME Noscapine | + | NAME Noscapine |
| + | ID COX00062 | ||
| + | FORMULA C22H23NO7 | ||
| + | EXACTMASS 413.147452095 | ||
| + | AVERAGEMASS 413.42056 | ||
| + | SMILES [H]C([H])([H])Oc(c([H])5)c(OC([H])([H])[H])c(C(=O)4)c(c([H])5)C([H])(O4)C([H])(N(C([H])([H])[H])1)c(c(OC([H])([H])[H])2)c(c([H])c(O3)c(OC([H])([H])3)2)C([H])([H])C([H])([H])1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
4544
CDK 9/16/09,17:8
53 57 0 0 0 0 0 0 0 0999 V2000
6.5678 0.1136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.5047 -0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.3170 -1.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.3170 -3.3000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9425 2.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8739 1.8634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9808 2.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8387 -1.9744 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.7448 -1.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7563 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6387 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8387 -3.0161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6387 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7448 -3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9584 0.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5047 -1.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9746 -1.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2800 1.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5047 -3.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3708 -1.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3708 -2.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2740 1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9776 -0.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6208 1.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3184 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6400 1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9006 -2.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3708 0.0048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2833 3.5299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.1933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2109 -1.1454 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3053 -0.7487 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2279 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.6297 -3.5998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3511 -4.0089 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1494 -3.9997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2867 -0.9354 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4389 -1.1590 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.6625 -2.0068 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5047 -4.1152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.7782 -0.6497 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.7103 0.5685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3615 -2.9100 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3615 -2.0805 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6808 -0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9077 0.3148 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0608 0.5417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7495 3.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8746 3.9961 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8171 3.1212 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.8792 2.8014 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3919 2.0724 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1208 1.5852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 10 1 0 0 0 0
1 22 1 0 0 0 0
2 16 1 0 0 0 0
2 28 1 0 0 0 0
3 20 1 0 0 0 0
3 27 1 0 0 0 0
4 21 1 0 0 0 0
4 27 1 0 0 0 0
5 24 1 0 0 0 0
5 29 1 0 0 0 0
6 22 2 0 0 0 0
7 26 1 0 0 0 0
7 30 1 0 0 0 0
8 9 1 0 0 0 0
8 12 1 0 0 0 0
8 17 1 0 0 0 0
9 10 1 0 0 0 0
9 11 1 0 0 0 0
9 31 1 0 0 0 0
10 15 1 0 0 0 0
10 32 1 0 0 0 0
11 13 1 0 0 0 0
11 16 2 0 0 0 0
12 14 1 0 0 0 0
12 33 1 0 0 0 0
12 34 1 0 0 0 0
13 14 1 0 0 0 0
13 19 2 0 0 0 0
14 35 1 0 0 0 0
14 36 1 0 0 0 0
15 18 2 0 0 0 0
15 23 1 0 0 0 0
16 20 1 0 0 0 0
17 37 1 0 0 0 0
17 38 1 0 0 0 0
17 39 1 0 0 0 0
18 22 1 0 0 0 0
18 24 1 0 0 0 0
19 21 1 0 0 0 0
19 40 1 0 0 0 0
20 21 2 0 0 0 0
23 25 2 0 0 0 0
23 41 1 0 0 0 0
24 26 2 0 0 0 0
25 26 1 0 0 0 0
25 42 1 0 0 0 0
27 43 1 0 0 0 0
27 44 1 0 0 0 0
28 45 1 0 0 0 0
28 46 1 0 0 0 0
28 47 1 0 0 0 0
29 48 1 0 0 0 0
29 49 1 0 0 0 0
29 50 1 0 0 0 0
30 51 1 0 0 0 0
30 52 1 0 0 0 0
30 53 1 0 0 0 0
S SKP 6
NAME Noscapine
ID COX00062
FORMULA C22H23NO7
EXACTMASS 413.147452095
AVERAGEMASS 413.42056
SMILES [H]C([H])([H])Oc(c([H])5)c(OC([H])([H])[H])c(C(=O)4)c(c([H])5)C([H])(O4)C([H])(N(C([H])([H])[H])1)c(c(OC([H])([H])[H])2)c(c([H])c(O3)c(OC([H])([H])3)2)C([H])([H])C([H])([H])1
M END