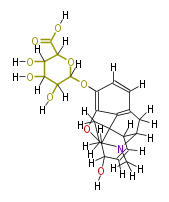
Mol:COX00057
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4318740 | + | 4318740 |
| − | CDK 9/16/09,17:7 | + | CDK 9/16/09,17:7 |
| − | + | ||
| − | 60 65 0 0 0 0 0 0 0 0999 V2000 | + | 60 65 0 0 0 0 0 0 0 0999 V2000 |
| − | 5.3664 -1.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3664 -1.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 -4.2472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 -4.2472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 0.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 0.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2690 2.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2690 2.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8671 1.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8671 1.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8671 3.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8671 3.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5369 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5369 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4030 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4030 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2956 -2.7961 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2956 -2.7961 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6483 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6483 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4720 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4720 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3742 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3742 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4720 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4720 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7268 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7268 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6483 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6483 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6483 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6483 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 -4.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 -4.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6483 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6483 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9768 -3.5282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9768 -3.5282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 0.5327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 0.5327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 1.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 1.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 2.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 2.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2690 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2690 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4030 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4030 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1853 -3.1061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1853 -3.1061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0089 -2.0105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0089 -2.0105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4055 -2.9685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4055 -2.9685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5110 -1.0184 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5110 -1.0184 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8145 -1.3563 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8145 -1.3563 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0826 -1.4771 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0826 -1.4771 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6840 -0.7869 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6840 -0.7869 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3190 -1.4393 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3190 -1.4393 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6339 -1.0101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6339 -1.0101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 -4.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 -4.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1853 -4.0572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1853 -4.0572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 -4.8427 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 -4.8427 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1853 0.3671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1853 0.3671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5229 -3.9505 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5229 -3.9505 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3991 -3.9821 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3991 -3.9821 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.4307 -3.1059 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.4307 -3.1059 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8247 1.1527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8247 1.1527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 -4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 -4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 1.2472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 1.2472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 1.4372 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 1.4372 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 3.6772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 3.6772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6720 3.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6720 3.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 2.7472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 2.7472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4040 1.8671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4040 1.8671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4040 3.2471 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4040 3.2471 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6720 4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6720 4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 3.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 3.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 14 1 0 0 0 0 | 1 14 1 0 0 0 0 | ||
1 22 1 0 0 0 0 | 1 22 1 0 0 0 0 | ||
| Line 128: | Line 128: | ||
32 33 1 0 0 0 0 | 32 33 1 0 0 0 0 | ||
32 56 1 0 0 0 0 | 32 56 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
| − | NAME Morphine_3_Glucuronide | + | NAME Morphine_3_Glucuronide |
| + | ID COX00057 | ||
| + | FORMULA C23H27NO9 | ||
| + | EXACTMASS 461.16858146699997 | ||
| + | AVERAGEMASS 461.46182 | ||
| + | SMILES [H]OC(=O)C([H])(O1)C([H])(O[H])C([H])(O[H])C([H])(O[H])C([H])(Oc(c([H])6)c(O5)c(c(c([H])6)2)C(C([H])([H])3)(C([H])54)C([H])(C([H])=C([H])C([H])(O[H])4)C([H])(N(C([H])([H])[H])C([H])([H])3)C([H])([H])2)1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
4318740
CDK 9/16/09,17:7
60 65 0 0 0 0 0 0 0 0999 V2000
5.3664 -1.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 -4.2472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 0.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2690 2.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8671 1.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8671 3.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5369 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4030 4.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.2956 -2.7961 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6483 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4720 -2.3205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 -2.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3742 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4720 -1.3695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7268 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6483 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6483 -3.7472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 -0.8939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 -4.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6483 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9768 -3.5282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 0.0571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 0.5327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 1.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 2.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2690 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4030 3.5572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1853 -3.1061 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.0089 -2.0105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4055 -2.9685 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5110 -1.0184 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8145 -1.3563 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.0826 -1.4771 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6840 -0.7869 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3190 -1.4393 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6339 -1.0101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 -4.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1853 -4.0572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 -4.8427 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1853 0.3671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.5229 -3.9505 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3991 -3.9821 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.4307 -3.1059 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8247 1.1527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 -4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.2472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 1.4372 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 3.6772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6720 3.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 2.7472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4040 1.8671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4040 3.2471 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6720 4.8672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 3.3672 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 14 1 0 0 0 0
1 22 1 0 0 0 0
2 20 1 0 0 0 0
2 51 1 0 0 0 0
3 26 1 0 0 0 0
3 28 1 0 0 0 0
4 28 1 0 0 0 0
4 32 1 0 0 0 0
5 29 1 0 0 0 0
5 57 1 0 0 0 0
6 30 1 0 0 0 0
6 58 1 0 0 0 0
7 31 1 0 0 0 0
7 59 1 0 0 0 0
8 33 1 0 0 0 0
8 60 1 0 0 0 0
9 33 2 0 0 0 0
10 13 1 0 0 0 0
10 18 1 0 0 0 0
10 25 1 0 0 0 0
11 12 1 0 0 0 0
11 14 1 0 0 0 0
11 15 1 0 0 0 0
11 16 1 0 0 0 0
12 13 1 0 0 0 0
12 21 1 0 0 0 0
12 34 1 0 0 0 0
13 17 1 0 0 0 0
13 35 1 0 0 0 0
14 20 1 0 0 0 0
14 36 1 0 0 0 0
15 18 1 0 0 0 0
15 37 1 0 0 0 0
15 38 1 0 0 0 0
16 19 2 0 0 0 0
16 22 1 0 0 0 0
17 19 1 0 0 0 0
17 39 1 0 0 0 0
17 40 1 0 0 0 0
18 41 1 0 0 0 0
18 42 1 0 0 0 0
19 24 1 0 0 0 0
20 23 1 0 0 0 0
20 43 1 0 0 0 0
21 23 2 0 0 0 0
21 44 1 0 0 0 0
22 26 2 0 0 0 0
23 45 1 0 0 0 0
24 27 2 0 0 0 0
24 46 1 0 0 0 0
25 47 1 0 0 0 0
25 48 1 0 0 0 0
25 49 1 0 0 0 0
26 27 1 0 0 0 0
27 50 1 0 0 0 0
28 29 1 0 0 0 0
28 52 1 0 0 0 0
29 30 1 0 0 0 0
29 53 1 0 0 0 0
30 31 1 0 0 0 0
30 54 1 0 0 0 0
31 32 1 0 0 0 0
31 55 1 0 0 0 0
32 33 1 0 0 0 0
32 56 1 0 0 0 0
S SKP 6
NAME Morphine_3_Glucuronide
ID COX00057
FORMULA C23H27NO9
EXACTMASS 461.16858146699997
AVERAGEMASS 461.46182
SMILES [H]OC(=O)C([H])(O1)C([H])(O[H])C([H])(O[H])C([H])(O[H])C([H])(Oc(c([H])6)c(O5)c(c(c([H])6)2)C(C([H])([H])3)(C([H])54)C([H])(C([H])=C([H])C([H])(O[H])4)C([H])(N(C([H])([H])[H])C([H])([H])3)C([H])([H])2)1
M END