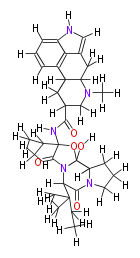
Mol:COX00036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 592735 | + | 592735 |
| − | CDK 9/16/09,17:2 | + | CDK 9/16/09,17:2 |
| − | + | ||
| − | 88 94 0 0 0 0 0 0 0 0999 V2000 | + | 88 94 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.9606 2.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9606 2.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3641 3.1790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3641 3.1790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1130 5.5122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1130 5.5122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4763 1.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4763 1.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3456 -0.8615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3456 -0.8615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3540 2.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3540 2.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5335 4.8016 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5335 4.8016 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2058 0.6418 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2058 0.6418 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8777 -0.8618 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8777 -0.8618 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8657 -5.0519 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8657 -5.0519 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3590 3.0783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3590 3.0783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9488 3.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9488 3.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9386 3.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9386 3.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7025 1.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7025 1.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9335 3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9335 3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0030 4.2963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0030 4.2963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5284 4.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5284 4.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5619 2.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5619 2.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9440 5.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9440 5.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 5.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 5.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0362 0.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0362 0.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9956 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9956 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8616 -1.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8616 -1.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0776 -0.8548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0776 -0.8548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0856 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0856 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9285 3.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9285 3.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9797 -0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9797 -0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0342 4.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0342 4.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8328 2.6933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8328 2.6933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2096 -0.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2096 -0.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7277 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7277 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9956 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9956 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0573 0.9644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0573 0.9644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3489 -0.1897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3489 -0.1897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8616 -3.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8616 -3.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7277 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7277 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5131 4.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5131 4.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7495 -0.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7495 -0.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8777 -4.9448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8777 -4.9448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0856 -3.9101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0856 -3.9101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3456 -4.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3456 -4.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9797 -5.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9797 -5.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0776 -4.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0776 -4.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3816 3.3572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3816 3.3572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4356 4.4785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4356 4.4785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3862 4.3596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3862 4.3596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8752 3.6896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8752 3.6896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4791 5.9152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4791 5.9152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6888 6.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6888 6.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8686 5.8963 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8686 5.8963 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3836 5.2234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3836 5.2234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8423 1.3492 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8423 1.3492 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2628 -2.8339 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2628 -2.8339 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6010 -1.4840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6010 -1.4840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3439 -1.2841 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3439 -1.2841 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4740 -1.7950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4740 -1.7950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8811 -2.4817 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8811 -2.4817 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7415 0.9539 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7415 0.9539 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7599 2.9909 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7599 2.9909 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4868 3.3179 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4868 3.3179 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3806 0.1389 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3806 0.1389 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5824 0.1420 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5824 0.1420 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6511 4.6207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6511 4.6207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0967 5.3000 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0967 5.3000 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4174 4.7456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4174 4.7456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2160 2.7557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2160 2.7557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7704 2.0764 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7704 2.0764 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4497 2.6309 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4497 2.6309 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0016 2.6760 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0016 2.6760 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3383 -2.5109 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3383 -2.5109 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9397 -1.8207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9397 -1.8207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1838 1.5713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1838 1.5713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4503 1.0910 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4503 1.0910 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9307 0.3575 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9307 0.3575 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7600 -0.3836 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7600 -0.3836 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5428 -0.7786 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5428 -0.7786 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9378 0.0042 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9378 0.0042 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0161 4.0363 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0161 4.0363 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8756 4.9018 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8756 4.9018 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0101 4.7613 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0101 4.7613 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0532 -0.9123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0532 -0.9123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2899 -0.0680 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2899 -0.0680 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4457 0.1687 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4457 0.1687 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5523 -3.5939 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5523 -3.5939 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9634 -4.1299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9634 -4.1299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1517 -5.6020 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1517 -5.6020 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9821 -6.0925 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9821 -6.0925 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5395 -5.2596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5395 -5.2596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 11 1 0 0 0 0 | 1 11 1 0 0 0 0 | ||
1 14 1 0 0 0 0 | 1 14 1 0 0 0 0 | ||
| Line 185: | Line 185: | ||
42 87 1 0 0 0 0 | 42 87 1 0 0 0 0 | ||
43 88 1 0 0 0 0 | 43 88 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
| − | NAME Ergoloid_Mesylate | + | NAME Ergoloid_Mesylate |
| + | ID COX00036 | ||
| + | FORMULA C33H45N5O5 | ||
| + | EXACTMASS 591.342069575 | ||
| + | AVERAGEMASS 591.7411 | ||
| + | SMILES C(C([H])([H])C([H])([H])[H])(C(C6=O)(N(C5(C(C7([H])[H])([H])N6C(C7([H])[H])([H])[H])O[H])C(=O)C(C([H])(C([H])([H])[H])C([H])([H])[H])(O5)N([H])C(C(C4([H])[H])([H])C(C(c31)(C(N4C([H])([H])[H])([H])C(c(c([H])2)c1c(c([H])c(c([H])3)[H])n([H])2)([H])[H])[H])([H])[H])=O)[H])(C([H])([H])[H])C([H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
592735
CDK 9/16/09,17:2
88 94 0 0 0 0 0 0 0 0999 V2000
2.9606 2.1676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3641 3.1790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1130 5.5122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4763 1.6007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3456 -0.8615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3540 2.9776 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.5335 4.8016 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.2058 0.6418 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.8777 -0.8618 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.8657 -5.0519 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.3590 3.0783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9488 3.9903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9386 3.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7025 1.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9335 3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0030 4.2963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5284 4.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5619 2.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9440 5.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 5.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0362 0.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9956 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8616 -1.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0776 -0.8548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0856 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9285 3.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9797 -0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0342 4.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8328 2.6933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2096 -0.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7277 -2.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9956 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0573 0.9644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3489 -0.1897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8616 -3.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7277 -3.4033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5131 4.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7495 -0.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8777 -4.9448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0856 -3.9101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3456 -4.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9797 -5.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0776 -4.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3816 3.3572 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4356 4.4785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3862 4.3596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.8752 3.6896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4791 5.9152 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.6888 6.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.8686 5.8963 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3836 5.2234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8423 1.3492 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.2628 -2.8339 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.6010 -1.4840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3439 -1.2841 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4740 -1.7950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.8811 -2.4817 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7415 0.9539 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7599 2.9909 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4868 3.3179 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3806 0.1389 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5824 0.1420 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.6511 4.6207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0967 5.3000 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4174 4.7456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.2160 2.7557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7704 2.0764 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4497 2.6309 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0016 2.6760 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.3383 -2.5109 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.9397 -1.8207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1838 1.5713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4503 1.0910 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.9307 0.3575 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.7600 -0.3836 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5428 -0.7786 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9378 0.0042 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0161 4.0363 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8756 4.9018 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0101 4.7613 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0532 -0.9123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.2899 -0.0680 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4457 0.1687 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5523 -3.5939 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9634 -4.1299 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1517 -5.6020 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9821 -6.0925 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5395 -5.2596 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 11 1 0 0 0 0
1 14 1 0 0 0 0
2 11 1 0 0 0 0
2 69 1 0 0 0 0
3 17 2 0 0 0 0
4 18 2 0 0 0 0
5 30 2 0 0 0 0
6 11 1 0 0 0 0
6 13 1 0 0 0 0
6 18 1 0 0 0 0
7 12 1 0 0 0 0
7 17 1 0 0 0 0
7 19 1 0 0 0 0
8 14 1 0 0 0 0
8 30 1 0 0 0 0
8 58 1 0 0 0 0
9 23 1 0 0 0 0
9 27 1 0 0 0 0
9 38 1 0 0 0 0
10 39 1 0 0 0 0
10 41 1 0 0 0 0
10 86 1 0 0 0 0
11 12 1 0 0 0 0
12 16 1 0 0 0 0
12 44 1 0 0 0 0
13 15 1 0 0 0 0
13 17 1 0 0 0 0
13 45 1 0 0 0 0
14 18 1 0 0 0 0
14 21 1 0 0 0 0
15 26 1 0 0 0 0
15 28 1 0 0 0 0
15 29 1 0 0 0 0
16 20 1 0 0 0 0
16 46 1 0 0 0 0
16 47 1 0 0 0 0
19 20 1 0 0 0 0
19 48 1 0 0 0 0
19 49 1 0 0 0 0
20 50 1 0 0 0 0
20 51 1 0 0 0 0
21 33 1 0 0 0 0
21 34 1 0 0 0 0
21 52 1 0 0 0 0
22 23 1 0 0 0 0
22 25 1 0 0 0 0
22 32 1 0 0 0 0
22 53 1 0 0 0 0
23 31 1 0 0 0 0
23 54 1 0 0 0 0
24 25 1 0 0 0 0
24 27 1 0 0 0 0
24 30 1 0 0 0 0
24 55 1 0 0 0 0
25 56 1 0 0 0 0
25 57 1 0 0 0 0
26 37 1 0 0 0 0
26 59 1 0 0 0 0
26 60 1 0 0 0 0
27 61 1 0 0 0 0
27 62 1 0 0 0 0
28 63 1 0 0 0 0
28 64 1 0 0 0 0
28 65 1 0 0 0 0
29 66 1 0 0 0 0
29 67 1 0 0 0 0
29 68 1 0 0 0 0
31 36 1 0 0 0 0
31 70 1 0 0 0 0
31 71 1 0 0 0 0
32 35 2 0 0 0 0
32 40 1 0 0 0 0
33 72 1 0 0 0 0
33 73 1 0 0 0 0
33 74 1 0 0 0 0
34 75 1 0 0 0 0
34 76 1 0 0 0 0
34 77 1 0 0 0 0
35 36 1 0 0 0 0
35 39 1 0 0 0 0
36 41 2 0 0 0 0
37 78 1 0 0 0 0
37 79 1 0 0 0 0
37 80 1 0 0 0 0
38 81 1 0 0 0 0
38 82 1 0 0 0 0
38 83 1 0 0 0 0
39 42 2 0 0 0 0
40 43 2 0 0 0 0
40 84 1 0 0 0 0
41 85 1 0 0 0 0
42 43 1 0 0 0 0
42 87 1 0 0 0 0
43 88 1 0 0 0 0
S SKP 6
NAME Ergoloid_Mesylate
ID COX00036
FORMULA C33H45N5O5
EXACTMASS 591.342069575
AVERAGEMASS 591.7411
SMILES C(C([H])([H])C([H])([H])[H])(C(C6=O)(N(C5(C(C7([H])[H])([H])N6C(C7([H])[H])([H])[H])O[H])C(=O)C(C([H])(C([H])([H])[H])C([H])([H])[H])(O5)N([H])C(C(C4([H])[H])([H])C(C(c31)(C(N4C([H])([H])[H])([H])C(c(c([H])2)c1c(c([H])c(c([H])3)[H])n([H])2)([H])[H])[H])([H])[H])=O)[H])(C([H])([H])[H])C([H])([H])[H]
M END