Mol:COX00019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 2476 | + | 2476 |
| − | CDK 9/16/09,16:59 | + | CDK 9/16/09,16:59 |
| − | + | ||
| − | 75 81 0 0 0 0 0 0 0 0999 V2000 | + | 75 81 0 0 0 0 0 0 0 0999 V2000 |
| − | 5.0395 -2.5677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0395 -2.5677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9653 -2.0758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9653 -2.0758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7551 -3.3197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7551 -3.3197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6200 -2.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6200 -2.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0154 1.4411 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0154 1.4411 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0154 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0154 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5328 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5328 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9805 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9805 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0154 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0154 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3478 -1.4095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3478 -1.4095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7124 -0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7124 -0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5328 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5328 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9805 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9805 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4631 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4631 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0503 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0503 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5677 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5677 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5677 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5677 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0514 -2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0514 -2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0395 1.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0395 1.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0851 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0851 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0851 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0851 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9220 1.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9220 1.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8477 -2.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8477 -2.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0098 2.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0098 2.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1624 -1.9070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1624 -1.9070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1200 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1200 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1200 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1200 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5840 3.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5840 3.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5878 3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5878 3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6081 -1.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6081 -1.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6374 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6374 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6439 -3.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6439 -3.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2425 -3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2425 -3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8465 -2.9215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8465 -2.9215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2660 -2.7144 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2660 -2.7144 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5592 -1.0924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5592 -1.0924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2565 -0.9776 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2565 -0.9776 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0294 -0.1474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0294 -0.1474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3828 0.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3828 0.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5631 -0.0185 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5631 -0.0185 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8729 0.3801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8729 0.3801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9380 -1.4649 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9380 -1.4649 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9380 -0.6678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9380 -0.6678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4668 -0.0207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4668 -0.0207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6487 -0.0683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6487 -0.0683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6754 1.2159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6754 1.2159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9851 0.8174 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9851 0.8174 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1109 2.2305 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1109 2.2305 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4205 1.6485 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4205 1.6485 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0821 1.2642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0821 1.2642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5397 1.9168 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5397 1.9168 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5923 2.6468 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5923 2.6468 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4461 -1.3557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4461 -1.3557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6111 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6111 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8786 -2.4582 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8786 -2.4582 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8100 0.3064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8100 0.3064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1504 -3.4566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1504 -3.4566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1457 3.4156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1457 3.4156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7448 4.2768 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7448 4.2768 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5342 4.3835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5342 4.3835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9888 3.6058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9888 3.6058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0831 -1.7083 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0831 -1.7083 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0067 -0.8348 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0067 -0.8348 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1332 -0.9112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1332 -0.9112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0174 -1.0664 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0174 -1.0664 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2687 -4.0684 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2687 -4.0684 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1375 -3.9499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1375 -3.9499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0190 -3.0812 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0190 -3.0812 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7489 -3.3907 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7489 -3.3907 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8674 -4.2595 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8674 -4.2595 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7361 -4.1410 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7361 -4.1410 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8764 -3.5408 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8764 -3.5408 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4658 -2.8916 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4658 -2.8916 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8166 -2.3022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8166 -2.3022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 9 1 0 0 0 0 | 1 9 1 0 0 0 0 | ||
1 21 1 0 0 0 0 | 1 21 1 0 0 0 0 | ||
| Line 159: | Line 159: | ||
34 73 1 0 0 0 0 | 34 73 1 0 0 0 0 | ||
34 74 1 0 0 0 0 | 34 74 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
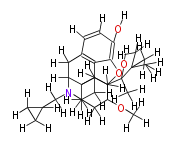
| − | NAME Buprenorphine | + | NAME Buprenorphine |
| + | ID COX00019 | ||
| + | FORMULA C29H41NO4 | ||
| + | EXACTMASS 467.30355880499997 | ||
| + | AVERAGEMASS 467.64017999999993 | ||
| + | SMILES C(O[H])(C(C([H])([H])[H])(C([H])([H])[H])C([H])([H])[H])(C(C([H])([H])6)([H])C(C([H])([H])7)(C(C514)(Oc(c(O[H])2)c1c(C(C(C56C7([H])[H])([H])N(C([H])([H])C4([H])[H])C([H])([H])C(C3([H])[H])(C3([H])[H])[H])([H])[H])c([H])c([H])2)[H])OC([H])([H])[H])C([H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
2476
CDK 9/16/09,16:59
75 81 0 0 0 0 0 0 0 0999 V2000
5.0395 -2.5677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.9653 -2.0758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7551 -3.3197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6200 -2.7682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0154 1.4411 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.0154 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5328 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9805 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0154 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3478 -1.4095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7124 -0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5328 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9805 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4631 -1.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0503 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5677 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5677 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0514 -2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0395 1.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0851 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0851 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9220 1.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8477 -2.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0098 2.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1624 -1.9070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1200 -0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1200 -1.9022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5840 3.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5878 3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.6081 -1.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6374 -1.0664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6439 -3.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2425 -3.7659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8465 -2.9215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2660 -2.7144 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5592 -1.0924 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2565 -0.9776 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0294 -0.1474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3828 0.6053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5631 -0.0185 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8729 0.3801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.9380 -1.4649 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.9380 -0.6678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4668 -0.0207 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6487 -0.0683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.6754 1.2159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9851 0.8174 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1109 2.2305 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4205 1.6485 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0821 1.2642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5397 1.9168 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5923 2.6468 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4461 -1.3557 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.6111 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.8786 -2.4582 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8100 0.3064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1504 -3.4566 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1457 3.4156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.7448 4.2768 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5342 4.3835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9888 3.6058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.0831 -1.7083 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.0067 -0.8348 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1332 -0.9112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0174 -1.0664 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2687 -4.0684 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1375 -3.9499 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0190 -3.0812 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7489 -3.3907 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8674 -4.2595 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7361 -4.1410 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8764 -3.5408 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4658 -2.8916 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8166 -2.3022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 9 1 0 0 0 0
1 21 1 0 0 0 0
2 8 1 0 0 0 0
2 30 1 0 0 0 0
3 18 1 0 0 0 0
3 57 1 0 0 0 0
4 27 1 0 0 0 0
4 75 1 0 0 0 0
5 12 1 0 0 0 0
5 19 1 0 0 0 0
5 22 1 0 0 0 0
6 7 1 0 0 0 0
6 11 1 0 0 0 0
6 12 1 0 0 0 0
6 13 1 0 0 0 0
7 9 1 0 0 0 0
7 15 1 0 0 0 0
7 16 1 0 0 0 0
8 9 1 0 0 0 0
8 10 1 0 0 0 0
8 14 1 0 0 0 0
9 35 1 0 0 0 0
10 11 1 0 0 0 0
10 18 1 0 0 0 0
10 36 1 0 0 0 0
11 37 1 0 0 0 0
11 38 1 0 0 0 0
12 17 1 0 0 0 0
12 39 1 0 0 0 0
13 14 1 0 0 0 0
13 40 1 0 0 0 0
13 41 1 0 0 0 0
14 42 1 0 0 0 0
14 43 1 0 0 0 0
15 19 1 0 0 0 0
15 44 1 0 0 0 0
15 45 1 0 0 0 0
16 20 2 0 0 0 0
16 21 1 0 0 0 0
17 20 1 0 0 0 0
17 46 1 0 0 0 0
17 47 1 0 0 0 0
18 23 1 0 0 0 0
18 25 1 0 0 0 0
19 48 1 0 0 0 0
19 49 1 0 0 0 0
20 26 1 0 0 0 0
21 27 2 0 0 0 0
22 24 1 0 0 0 0
22 50 1 0 0 0 0
22 51 1 0 0 0 0
23 32 1 0 0 0 0
23 33 1 0 0 0 0
23 34 1 0 0 0 0
24 28 1 0 0 0 0
24 29 1 0 0 0 0
24 52 1 0 0 0 0
25 53 1 0 0 0 0
25 54 1 0 0 0 0
25 55 1 0 0 0 0
26 31 2 0 0 0 0
26 56 1 0 0 0 0
27 31 1 0 0 0 0
28 29 1 0 0 0 0
28 58 1 0 0 0 0
28 59 1 0 0 0 0
29 60 1 0 0 0 0
29 61 1 0 0 0 0
30 62 1 0 0 0 0
30 63 1 0 0 0 0
30 64 1 0 0 0 0
31 65 1 0 0 0 0
32 66 1 0 0 0 0
32 67 1 0 0 0 0
32 68 1 0 0 0 0
33 69 1 0 0 0 0
33 70 1 0 0 0 0
33 71 1 0 0 0 0
34 72 1 0 0 0 0
34 73 1 0 0 0 0
34 74 1 0 0 0 0
S SKP 6
NAME Buprenorphine
ID COX00019
FORMULA C29H41NO4
EXACTMASS 467.30355880499997
AVERAGEMASS 467.64017999999993
SMILES C(O[H])(C(C([H])([H])[H])(C([H])([H])[H])C([H])([H])[H])(C(C([H])([H])6)([H])C(C([H])([H])7)(C(C514)(Oc(c(O[H])2)c1c(C(C(C56C7([H])[H])([H])N(C([H])([H])C4([H])[H])C([H])([H])C(C3([H])[H])(C3([H])[H])[H])([H])[H])c([H])c([H])2)[H])OC([H])([H])[H])C([H])([H])[H]
M END