Mol:COX00005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 197 | + | 197 |
| − | CDK 9/16/09,16:56 | + | CDK 9/16/09,16:56 |
| − | + | ||
| − | 42 44 0 0 0 0 0 0 0 0999 V2000 | + | 42 44 0 0 0 0 0 0 0 0999 V2000 |
| − | 8.4752 2.2720 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4752 2.2720 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1980 2.4500 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1980 2.4500 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9405 0.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9405 0.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4026 1.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4026 1.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6844 2.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6844 2.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6651 1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6651 1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2852 2.8584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2852 2.8584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0615 1.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0615 1.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8888 3.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8888 3.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.1109 2.0417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.1109 2.0417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6063 3.3629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6063 3.3629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7897 1.5372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7897 1.5372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6783 -0.7504 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6783 -0.7504 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6783 -2.3598 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6783 -2.3598 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -0.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -0.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.0551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.0551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -3.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -3.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4026 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4026 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9917 1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9917 1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9889 0.2002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9889 0.2002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9422 1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9422 1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7523 2.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7523 2.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2619 -1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2619 -1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -2.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -2.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0180 1.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0180 1.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5938 2.4182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5938 2.4182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4266 -0.2390 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4266 -0.2390 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6989 1.1203 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6989 1.1203 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0999 2.6073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0999 2.6073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3070 2.5254 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3070 2.5254 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0935 1.5494 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0935 1.5494 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1000 3.2298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1000 3.2298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8819 -1.5551 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8819 -1.5551 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -0.7451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -0.7451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3291 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3291 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4030 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4030 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8084 0.8960 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8084 0.8960 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6131 2.4053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6131 2.4053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2428 3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2428 3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 6 1 0 0 0 0 | 1 6 1 0 0 0 0 | ||
1 7 1 0 0 0 0 | 1 7 1 0 0 0 0 | ||
| Line 89: | Line 89: | ||
25 26 2 0 0 0 0 | 25 26 2 0 0 0 0 | ||
27 37 1 0 0 0 0 | 27 37 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
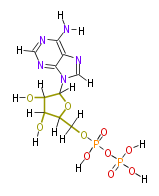
| − | NAME Adenosine_Diphosphate | + | NAME Adenosine_Diphosphate |
| + | ID COX00005 | ||
| + | FORMULA C10H15N5O10P2 | ||
| + | EXACTMASS 427.029414745 | ||
| + | AVERAGEMASS 427.201322 | ||
| + | SMILES [H]OC([H])(C([H])(O[H])1)C([H])(OC([H])(n(c([H])3)c(n2)c(n3)c(N([H])[H])nc([H])2)1)C([H])([H])OP(=O)(O[H])OP(=O)(O[H])O[H] | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
197
CDK 9/16/09,16:56
42 44 0 0 0 0 0 0 0 0999 V2000
8.4752 2.2720 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
10.1980 2.4500 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
5.9405 0.5075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4026 1.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6844 2.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.6651 1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.2852 2.8584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.0615 1.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.8888 3.0820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.1109 2.0417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.6063 3.3629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.7897 1.5372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6783 -0.7504 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.6783 -2.3598 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -0.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.0551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -3.5551 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.4026 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9917 1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9889 0.2002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9422 1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7523 2.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2619 -1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -2.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0180 1.7682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5938 2.4182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4266 -0.2390 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.6989 1.1203 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0999 2.6073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3070 2.5254 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.0935 1.5494 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1000 3.2298 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8819 -1.5551 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -0.7451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3291 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4030 -3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.8084 0.8960 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.6131 2.4053 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.2428 3.8651 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 6 1 0 0 0 0
1 7 1 0 0 0 0
1 8 1 0 0 0 0
1 9 2 0 0 0 0
2 7 1 0 0 0 0
2 10 1 0 0 0 0
2 11 1 0 0 0 0
2 12 2 0 0 0 0
3 20 1 0 0 0 0
3 21 1 0 0 0 0
4 18 1 0 0 0 0
4 34 1 0 0 0 0
5 19 1 0 0 0 0
5 35 1 0 0 0 0
6 22 1 0 0 0 0
8 40 1 0 0 0 0
10 41 1 0 0 0 0
11 42 1 0 0 0 0
13 20 1 0 0 0 0
13 23 1 0 0 0 0
13 24 1 0 0 0 0
14 24 2 0 0 0 0
14 25 1 0 0 0 0
15 23 2 0 0 0 0
15 27 1 0 0 0 0
16 26 1 0 0 0 0
16 27 2 0 0 0 0
17 26 1 0 0 0 0
17 38 1 0 0 0 0
17 39 1 0 0 0 0
18 19 1 0 0 0 0
18 20 1 0 0 0 0
18 28 1 0 0 0 0
19 21 1 0 0 0 0
19 29 1 0 0 0 0
20 30 1 0 0 0 0
21 22 1 0 0 0 0
21 31 1 0 0 0 0
22 32 1 0 0 0 0
22 33 1 0 0 0 0
23 25 1 0 0 0 0
24 36 1 0 0 0 0
25 26 2 0 0 0 0
27 37 1 0 0 0 0
S SKP 6
NAME Adenosine_Diphosphate
ID COX00005
FORMULA C10H15N5O10P2
EXACTMASS 427.029414745
AVERAGEMASS 427.201322
SMILES [H]OC([H])(C([H])(O[H])1)C([H])(OC([H])(n(c([H])3)c(n2)c(n3)c(N([H])[H])nc([H])2)1)C([H])([H])OP(=O)(O[H])OP(=O)(O[H])O[H]
M END