Mol:COX00002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4159 | + | 4159 |
| − | CDK 9/16/09,16:56 | + | CDK 9/16/09,16:56 |
| − | + | ||
| − | 57 60 0 0 0 0 0 0 0 0999 V2000 | + | 57 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 8.0319 2.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0319 2.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7950 1.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7950 1.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0465 2.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0465 2.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8722 2.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8722 2.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.9606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.9606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3931 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3931 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3931 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3931 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5271 -0.4083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5271 -0.4083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6610 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6610 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3393 1.3965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3393 1.3965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5271 1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5271 1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6610 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6610 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3393 -0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3393 -0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7510 -0.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7510 -0.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9229 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9229 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5431 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5431 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6451 -1.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6451 -1.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3931 2.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3931 2.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7430 -1.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7430 -1.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1493 1.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1493 1.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7587 0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7587 0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8242 0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8242 0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6489 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6489 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8076 -2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8076 -2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0622 1.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0622 1.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8763 -0.3790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8763 -0.3790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8680 -1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8680 -1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4830 -0.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4830 -0.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2664 -0.8276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2664 -0.8276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3972 0.5167 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3972 0.5167 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9256 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9256 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1285 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1285 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6610 1.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6610 1.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0883 -0.7799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0883 -0.7799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8767 -0.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8767 -0.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3838 0.1770 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3838 0.1770 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3838 1.0065 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3838 1.0065 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7612 -2.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7612 -2.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1523 -1.3343 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1523 -1.3343 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9098 -2.4040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9098 -2.4040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0131 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0131 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3931 2.7117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3931 2.7117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7731 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7731 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3787 0.5801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3787 0.5801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7635 1.2048 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7635 1.2048 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1388 0.5896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1388 0.5896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8361 0.7688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8361 0.7688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4476 2.8081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4476 2.8081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7950 2.2117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7950 2.2117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0289 -2.9799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0289 -2.9799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6513 -3.5975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6513 -3.5975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2689 -2.9752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2689 -2.9752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8100 -2.6265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8100 -2.6265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7146 1.0611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7146 1.0611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5074 1.1431 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5074 1.1431 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3430 -0.0628 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3430 -0.0628 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4382 1.9078 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4382 1.9078 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 10 1 0 0 0 0 | 1 10 1 0 0 0 0 | ||
1 48 1 0 0 0 0 | 1 48 1 0 0 0 0 | ||
| Line 120: | Line 120: | ||
26 27 1 0 0 0 0 | 26 27 1 0 0 0 0 | ||
26 56 1 0 0 0 0 | 26 56 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
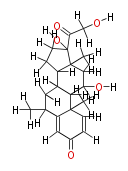
| − | NAME 6a_Methylprednisolone | + | NAME 6a_Methylprednisolone |
| + | ID COX00002 | ||
| + | FORMULA C22H30O5 | ||
| + | EXACTMASS 374.20932407 | ||
| + | AVERAGEMASS 374.47060000000005 | ||
| + | SMILES [H]OC([H])([H])C(=O)C(O[H])(C([H])([H])4)C(C([H])([H])[H])(C([H])([H])3)C([H])(C([H])([H])4)C([H])(C([H])([H])1)C([H])(C([H])(O[H])3)C(C([H])=2)(C([H])([H])[H])C(=C([H])C(=O)C([H])2)C([H])(C([H])([H])[H])1 | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
4159
CDK 9/16/09,16:56
57 60 0 0 0 0 0 0 0 0999 V2000
8.0319 2.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7950 1.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.0465 2.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.8722 2.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.9606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.3931 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3931 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5271 -0.4083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6610 0.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3393 1.3965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5271 1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6610 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3393 -0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7510 -0.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9229 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5431 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6451 -1.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3931 2.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7430 -1.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1493 1.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7587 0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8242 0.1490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6489 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8076 -2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0622 1.5745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8763 -0.3790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8680 -1.4640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4830 -0.7535 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2664 -0.8276 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3972 0.5167 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9256 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1285 2.0667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6610 1.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0883 -0.7799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.8767 -0.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3838 0.1770 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3838 1.0065 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7612 -2.0302 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.1523 -1.3343 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9098 -2.4040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0131 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.3931 2.7117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7731 2.0917 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3787 0.5801 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7635 1.2048 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.1388 0.5896 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8361 0.7688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4476 2.8081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7950 2.2117 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0289 -2.9799 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6513 -3.5975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.2689 -2.9752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8100 -2.6265 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.7146 1.0611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.5074 1.1431 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3430 -0.0628 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.4382 1.9078 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 10 1 0 0 0 0
1 48 1 0 0 0 0
2 12 1 0 0 0 0
2 49 1 0 0 0 0
3 20 2 0 0 0 0
4 25 1 0 0 0 0
4 57 1 0 0 0 0
5 27 2 0 0 0 0
6 7 1 0 0 0 0
6 10 1 0 0 0 0
6 11 1 0 0 0 0
6 18 1 0 0 0 0
7 8 1 0 0 0 0
7 13 1 0 0 0 0
7 28 1 0 0 0 0
8 9 1 0 0 0 0
8 16 1 0 0 0 0
8 29 1 0 0 0 0
9 12 1 0 0 0 0
9 14 1 0 0 0 0
9 30 1 0 0 0 0
10 15 1 0 0 0 0
10 20 1 0 0 0 0
11 12 1 0 0 0 0
11 31 1 0 0 0 0
11 32 1 0 0 0 0
12 33 1 0 0 0 0
13 15 1 0 0 0 0
13 34 1 0 0 0 0
13 35 1 0 0 0 0
14 19 1 0 0 0 0
14 21 1 0 0 0 0
14 22 1 0 0 0 0
15 36 1 0 0 0 0
15 37 1 0 0 0 0
16 17 1 0 0 0 0
16 38 1 0 0 0 0
16 39 1 0 0 0 0
17 19 1 0 0 0 0
17 23 1 0 0 0 0
17 40 1 0 0 0 0
18 41 1 0 0 0 0
18 42 1 0 0 0 0
18 43 1 0 0 0 0
19 24 2 0 0 0 0
20 25 1 0 0 0 0
21 44 1 0 0 0 0
21 45 1 0 0 0 0
21 46 1 0 0 0 0
22 26 2 0 0 0 0
22 47 1 0 0 0 0
23 50 1 0 0 0 0
23 51 1 0 0 0 0
23 52 1 0 0 0 0
24 27 1 0 0 0 0
24 53 1 0 0 0 0
25 54 1 0 0 0 0
25 55 1 0 0 0 0
26 27 1 0 0 0 0
26 56 1 0 0 0 0
S SKP 6
NAME 6a_Methylprednisolone
ID COX00002
FORMULA C22H30O5
EXACTMASS 374.20932407
AVERAGEMASS 374.47060000000005
SMILES [H]OC([H])([H])C(=O)C(O[H])(C([H])([H])4)C(C([H])([H])[H])(C([H])([H])3)C([H])(C([H])([H])4)C([H])(C([H])([H])1)C([H])(C([H])(O[H])3)C(C([H])=2)(C([H])([H])[H])C(=C([H])C(=O)C([H])2)C([H])(C([H])([H])[H])1
M END