Mol:BMMCHC--a013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 58 0 0 1 0 0 0 0 0999 V2000 | + | 55 58 0 0 1 0 0 0 0 0999 V2000 |
| − | 2.8660 -2.9578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -2.9578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6092 -3.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6092 -3.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2024 -4.5405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2024 -4.5405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2079 -4.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2079 -4.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5873 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5873 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8963 -4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8963 -4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9444 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9444 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.0784 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.0784 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.0784 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.0784 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9444 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9444 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8105 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8105 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8105 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8105 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5536 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5536 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.1469 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.1469 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1523 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1523 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6765 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6765 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4832 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.4832 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 21.6911 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.6911 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.8251 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 20.8251 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.0820 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 20.0820 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 19.1038 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1038 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6047 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6047 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7206 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7206 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.4887 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.4887 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.4347 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.4347 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.5296 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5296 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1174 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1174 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.9418 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.9418 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.3386 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.3386 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.4565 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4565 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2486 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2486 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6644 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6644 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.4784 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.4784 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.8092 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.8092 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5524 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5524 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.0661 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.0661 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.1401 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1401 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.1620 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.1620 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4928 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4928 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2360 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2360 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.7497 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.7497 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8237 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 12.8237 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 11.8456 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8456 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.1764 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.1764 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1983 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1983 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5292 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5292 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5510 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5510 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8819 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8819 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9037 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9037 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2346 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2346 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2564 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2564 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1327 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1327 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5365 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5365 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2420 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2420 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8 13 2 0 0 0 0 | + | 8 13 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 12 11 2 0 0 0 0 | + | 12 11 2 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 14 12 1 0 0 0 0 | + | 14 12 1 0 0 0 0 |
| − | 11 16 1 0 0 0 0 | + | 11 16 1 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 21 25 1 6 0 0 0 | + | 21 25 1 6 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 18 25 1 6 0 0 0 | + | 18 25 1 6 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 18 16 1 0 0 0 0 | + | 18 16 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 38 35 1 0 0 0 0 | + | 38 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 35 34 1 0 0 0 0 | + | 35 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 43 53 1 1 0 0 0 | + | 43 53 1 1 0 0 0 |
| − | 44 54 2 0 0 0 0 | + | 44 54 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 48 55 2 0 0 0 0 | + | 48 55 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 19 23 1 1 0 0 0 | + | 19 23 1 1 0 0 0 |
| − | 20 24 1 1 0 0 0 | + | 20 24 1 1 0 0 0 |
| − | 28 27 1 0 0 0 0 | + | 28 27 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 27 29 2 0 0 0 0 | + | 27 29 2 0 0 0 0 |
| − | 24 27 1 0 0 0 0 | + | 24 27 1 0 0 0 0 |
| − | 52 6 1 0 0 0 0 | + | 52 6 1 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 5 4 2 0 0 0 0 | + | 5 4 2 0 0 0 0 |
| − | 3 2 2 0 0 0 0 | + | 3 2 2 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 1 5 1 0 0 0 0 | + | 1 5 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMMCHC--a013 | + | ID BMMCHC--a013 |
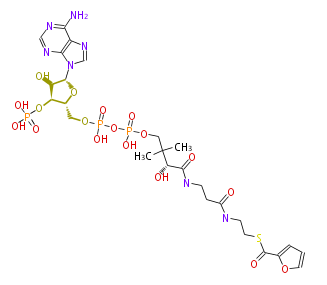
| − | NAME 2-Furoyl-CoA | + | NAME 2-Furoyl-CoA |
| − | FORMULA C26H38N7O18P3S | + | FORMULA C26H38N7O18P3S |
| − | EXACTMASS 861.1206 | + | EXACTMASS 861.1206 |
| − | AVERAGEMASS 861.6035 | + | AVERAGEMASS 861.6035 |
| − | SMILES C([C@H](C(NCCC(NCCSC(c(c4)occ4)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O | + | SMILES C([C@H](C(NCCC(NCCSC(c(c4)occ4)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00845 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00845 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 58 0 0 1 0 0 0 0 0999 V2000
2.8660 -2.9578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6092 -3.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2024 -4.5405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2079 -4.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5873 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8963 -4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.9444 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.0784 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.0784 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.9444 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.8105 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.8105 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.5536 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.1469 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.1523 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.6765 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.4832 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
21.6911 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.8251 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.0820 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
19.1038 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.6047 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.7206 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.4887 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.4347 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.5296 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
22.1174 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.9418 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.3386 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.4565 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
17.2486 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.6644 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.4784 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.8092 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
16.5524 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.0661 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.1401 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.1620 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.4928 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2360 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.7497 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.8237 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
11.8456 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.1764 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.1983 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.5292 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5510 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8819 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.9037 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2346 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2564 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
13.1327 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.5365 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.2420 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8 13 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
12 13 1 0 0 0 0
12 11 2 0 0 0 0
16 15 1 0 0 0 0
15 14 2 0 0 0 0
14 12 1 0 0 0 0
11 16 1 0 0 0 0
13 17 1 0 0 0 0
21 25 1 6 0 0 0
20 21 1 0 0 0 0
18 25 1 6 0 0 0
20 19 1 0 0 0 0
18 19 1 0 0 0 0
21 22 1 0 0 0 0
22 26 1 0 0 0 0
18 16 1 0 0 0 0
26 31 1 0 0 0 0
31 33 1 0 0 0 0
31 32 2 0 0 0 0
31 34 1 0 0 0 0
38 35 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
38 39 1 0 0 0 0
35 34 1 0 0 0 0
39 40 1 0 0 0 0
40 43 1 0 0 0 0
40 41 1 0 0 0 0
40 42 1 0 0 0 0
43 44 1 0 0 0 0
43 53 1 1 0 0 0
44 54 2 0 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
48 55 2 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
19 23 1 1 0 0 0
20 24 1 1 0 0 0
28 27 1 0 0 0 0
27 30 1 0 0 0 0
27 29 2 0 0 0 0
24 27 1 0 0 0 0
52 6 1 0 0 0 0
6 2 1 0 0 0 0
6 7 2 0 0 0 0
5 4 2 0 0 0 0
3 2 2 0 0 0 0
2 1 1 0 0 0 0
1 5 1 0 0 0 0
4 3 1 0 0 0 0
S SKP 7
ID BMMCHC--a013
NAME 2-Furoyl-CoA
FORMULA C26H38N7O18P3S
EXACTMASS 861.1206
AVERAGEMASS 861.6035
SMILES C([C@H](C(NCCC(NCCSC(c(c4)occ4)=O)=O)=O)O)(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)3)O[C@H]([C@@H]3O)n(c21)cnc(c(N)ncn2)1)(O)=O)(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00845
M END