Mol:BMFYB4ESa011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 60 0 0 1 0 0 0 0 0999 V2000 | + | 58 60 0 0 1 0 0 0 0 0999 V2000 |
| − | 6.0377 -3.4035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0377 -3.4035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0596 -3.1956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0596 -3.1956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3904 -3.9388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.3904 -3.9388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.4123 -3.7309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4123 -3.7309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7431 -4.4740 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0 | + | 2.7431 -4.4740 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4863 -5.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4863 -5.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0740 -5.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0740 -5.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6994 -4.8898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6994 -4.8898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3467 -4.3546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3467 -4.3546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.3948 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.3948 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.5288 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.5288 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.5288 -0.8736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.5288 -0.8736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.3948 -0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.3948 -0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2608 -0.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2608 -0.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2608 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2608 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0040 -0.2045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0040 -0.2045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.5972 0.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.5972 0.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6027 0.6045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6027 0.6045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.1269 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.1269 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9336 1.3477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 22.9336 1.3477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.1415 2.3258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 23.1415 2.3258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 22.2755 2.8258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 22.2755 2.8258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 21.5323 2.1567 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.5323 2.1567 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.5542 2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5542 2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0551 2.7326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0551 2.7326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1710 3.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1710 3.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9391 1.2432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9391 1.2432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8851 1.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8851 1.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9800 4.4081 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9800 4.4081 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5678 3.5991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5678 3.5991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.3922 5.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.3922 5.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.7890 4.9959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.7890 4.9959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.9069 1.8294 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.9069 1.8294 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6990 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6990 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.1148 2.8075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1148 2.8075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.9288 2.0373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.9288 2.0373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2596 1.2941 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2596 1.2941 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.0028 0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.0028 0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5165 1.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5165 1.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5905 0.5510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5905 0.5510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6124 0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.6124 0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9432 0.0158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9432 0.0158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6864 -0.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.6864 -0.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2001 0.6849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2001 0.6849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2741 -0.7274 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 14.2741 -0.7274 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 13.2959 -0.5195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.2959 -0.5195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6268 -1.2626 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.6268 -1.2626 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6487 -1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6487 -1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9795 -1.7978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9795 -1.7978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0014 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0014 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3323 -2.3331 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3323 -2.3331 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3541 -2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3541 -2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6850 -2.8683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6850 -2.8683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7068 -2.6604 0.0000 S 0 3 0 0 0 0 0 0 0 0 0 0 | + | 6.7068 -2.6604 0.0000 S 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5831 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5831 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9869 0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9869 0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6924 -0.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6924 -0.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11 16 2 0 0 0 0 | + | 11 16 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 19 18 1 0 0 0 0 | + | 19 18 1 0 0 0 0 |
| − | 18 17 2 0 0 0 0 | + | 18 17 2 0 0 0 0 |
| − | 17 15 1 0 0 0 0 | + | 17 15 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 24 28 1 6 0 0 0 | + | 24 28 1 6 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 21 28 1 6 0 0 0 | + | 21 28 1 6 0 0 0 |
| − | 23 22 1 0 0 0 0 | + | 23 22 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 29 34 1 0 0 0 0 | + | 29 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 34 36 2 0 0 0 0 | + | 34 36 2 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 38 37 1 0 0 0 0 | + | 38 37 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 56 1 1 0 0 0 | + | 46 56 1 1 0 0 0 |
| − | 47 57 2 0 0 0 0 | + | 47 57 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 58 2 0 0 0 0 | + | 51 58 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 22 26 1 1 0 0 0 | + | 22 26 1 1 0 0 0 |
| − | 23 27 1 1 0 0 0 | + | 23 27 1 1 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 30 32 2 0 0 0 0 | + | 30 32 2 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 55 1 1 0 0 0 0 | + | 55 1 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 1 10 2 0 0 0 0 | + | 1 10 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 3 9 1 6 0 0 0 | + | 3 9 1 6 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 5 8 1 0 0 0 0 | + | 5 8 1 0 0 0 0 |
| − | S SKP 6 | + | S SKP 6 |
| − | ID BMFYB4ESa011 | + | ID BMFYB4ESa011 |
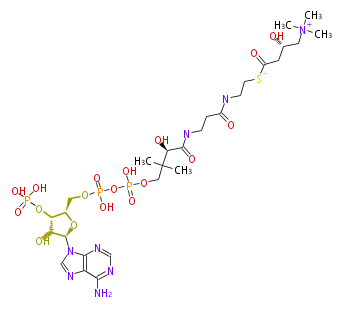
| − | NAME L-Carnitinyl-CoA | + | NAME L-Carnitinyl-CoA |
| − | FORMULA C28H51N8O18P3S | + | FORMULA C28H51N8O18P3S |
| − | EXACTMASS 912.2254 | + | EXACTMASS 912.2254 |
| − | AVERAGEMASS 912.7349 | + | AVERAGEMASS 912.7349 |
| − | SMILES [SH+1](C(=O)C[C@@H](C[N+1](C)(C)C)O)CCNC(CCNC(=O)[C@@H](C(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O)O)=O | + | SMILES [SH+1](C(=O)C[C@@H](C[N+1](C)(C)C)O)CCNC(CCNC(=O)[C@@H](C(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 60 0 0 1 0 0 0 0 0999 V2000
6.0377 -3.4035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0596 -3.1956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3904 -3.9388 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.4123 -3.7309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7431 -4.4740 0.0000 N 0 3 0 0 0 0 0 0 0 0 0 0
3.4863 -5.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0740 -5.2172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6994 -4.8898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3467 -4.3546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.3948 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.5288 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.5288 -0.8736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.3948 -0.3736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.2608 -0.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.2608 -1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.0040 -0.2045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.5972 0.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.6027 0.6045 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
25.1269 -2.3736 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.9336 1.3477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.1415 2.3258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
22.2755 2.8258 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
21.5323 2.1567 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.5542 2.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.0551 2.7326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.1710 3.8204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.9391 1.2432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.8851 1.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.9800 4.4081 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
23.5678 3.5991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.3922 5.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.7890 4.9959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.9069 1.8294 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
18.6990 0.8512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.1148 2.8075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.9288 2.0373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.2596 1.2941 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
18.0028 0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.5165 1.9633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.5905 0.5510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.6124 0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.9432 0.0158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.6864 -0.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2001 0.6849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2741 -0.7274 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
13.2959 -0.5195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.6268 -1.2626 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
11.6487 -1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.9795 -1.7978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0014 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.3323 -2.3331 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.3541 -2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6850 -2.8683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7068 -2.6604 0.0000 S 0 3 0 0 0 0 0 0 0 0 0 0
14.5831 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.9869 0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.6924 -0.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11 16 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
15 16 1 0 0 0 0
15 14 2 0 0 0 0
19 18 1 0 0 0 0
18 17 2 0 0 0 0
17 15 1 0 0 0 0
14 19 1 0 0 0 0
16 20 1 0 0 0 0
24 28 1 6 0 0 0
23 24 1 0 0 0 0
21 28 1 6 0 0 0
23 22 1 0 0 0 0
21 22 1 0 0 0 0
24 25 1 0 0 0 0
25 29 1 0 0 0 0
21 19 1 0 0 0 0
29 34 1 0 0 0 0
34 35 1 0 0 0 0
34 36 2 0 0 0 0
34 37 1 0 0 0 0
41 38 1 0 0 0 0
38 39 2 0 0 0 0
38 40 1 0 0 0 0
41 42 1 0 0 0 0
38 37 1 0 0 0 0
42 43 1 0 0 0 0
43 46 1 0 0 0 0
43 44 1 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
46 56 1 1 0 0 0
47 57 2 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
51 58 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
22 26 1 1 0 0 0
23 27 1 1 0 0 0
31 30 1 0 0 0 0
30 33 1 0 0 0 0
30 32 2 0 0 0 0
27 30 1 0 0 0 0
55 1 1 0 0 0 0
1 2 1 0 0 0 0
1 10 2 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
3 9 1 6 0 0 0
4 5 1 0 0 0 0
5 7 1 0 0 0 0
5 6 1 0 0 0 0
5 8 1 0 0 0 0
S SKP 6
ID BMFYB4ESa011
NAME L-Carnitinyl-CoA
FORMULA C28H51N8O18P3S
EXACTMASS 912.2254
AVERAGEMASS 912.7349
SMILES [SH+1](C(=O)C[C@@H](C[N+1](C)(C)C)O)CCNC(CCNC(=O)[C@@H](C(C)(C)COP(OP(OC[C@H]([C@@H](OP(O)(O)=O)1)O[C@@H](n(c23)cnc2c(ncn3)N)[C@@H]1O)(O)=O)(O)=O)O)=O
M END