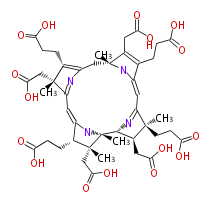
Mol:BMCCPPPC0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 68 0 0 1 0 0 0 0 0999 V2000 | + | 64 68 0 0 1 0 0 0 0 0999 V2000 |
| − | 7.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 7.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 6.3666 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 6.3666 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 5.3884 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.3884 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.7620 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7620 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4530 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4530 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5575 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5575 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9530 -1.7759 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.9530 -1.7759 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 5.7620 -2.6744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 5.7620 -2.6744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 6.8666 -2.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8666 -2.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8447 -2.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8447 -2.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8228 -2.1827 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 8.8228 -2.1827 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 9.9274 -2.6744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9274 -2.6744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7364 -1.7759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7364 -1.7759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1319 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1319 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2364 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2364 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9274 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9274 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3010 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 10.3010 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 9.3228 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 9.3228 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 8.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 8.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 6.4312 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4312 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0575 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0575 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6319 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6319 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2582 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2582 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1537 2.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1537 2.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9543 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9543 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7788 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7788 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1855 4.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1855 4.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4373 2.6756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4373 2.6756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6942 2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6942 2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7431 2.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7431 2.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0870 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0870 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2099 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2099 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2588 -2.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2588 -2.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5541 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5541 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6031 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6031 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3951 -4.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3951 -4.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1537 -2.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1537 -2.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1353 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1353 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0863 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0863 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9044 -2.0038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9044 -2.0038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3112 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3112 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3057 -3.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3057 -3.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2520 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2520 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8888 3.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8888 3.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8833 3.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8833 3.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.4711 3.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.4711 3.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3228 4.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3228 4.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1889 4.5773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1889 4.5773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8935 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8935 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7124 -3.9354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7124 -3.9354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1383 -5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1383 -5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4441 -5.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4441 -5.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0509 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0509 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5157 -2.8052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5157 -2.8052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5352 3.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5352 3.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 1.6464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 1.6464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8295 -3.2925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8295 -3.2925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2943 -4.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2943 -4.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.0643 4.7937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.0643 4.7937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4656 3.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4656 3.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0549 4.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0549 4.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1889 5.5773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1889 5.5773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1800 4.9044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1800 4.9044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5977 5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5977 5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17 44 1 1 0 0 0 | + | 17 44 1 1 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 36 51 1 0 0 0 0 | + | 36 51 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 36 52 2 0 0 0 0 | + | 36 52 2 0 0 0 0 |
| − | 11 22 1 6 0 0 0 | + | 11 22 1 6 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 33 53 1 0 0 0 0 | + | 33 53 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 33 54 2 0 0 0 0 | + | 33 54 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 16 2 0 0 0 0 | + | 23 16 2 0 0 0 0 |
| − | 30 55 1 0 0 0 0 | + | 30 55 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 30 56 2 0 0 0 0 | + | 30 56 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 19 23 1 1 0 0 0 | + | 19 23 1 1 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 15 14 2 0 0 0 0 | + | 15 14 2 0 0 0 0 |
| − | 12 38 1 0 0 0 0 | + | 12 38 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 39 57 1 0 0 0 0 | + | 39 57 1 0 0 0 0 |
| − | 10 9 1 0 0 0 0 | + | 10 9 1 0 0 0 0 |
| − | 39 58 2 0 0 0 0 | + | 39 58 2 0 0 0 0 |
| − | 6 21 2 0 0 0 0 | + | 6 21 2 0 0 0 0 |
| − | 7 31 1 1 0 0 0 | + | 7 31 1 1 0 0 0 |
| − | 3 28 1 1 0 0 0 | + | 3 28 1 1 0 0 0 |
| − | 46 59 1 0 0 0 0 | + | 46 59 1 0 0 0 0 |
| − | 9 8 2 0 0 0 0 | + | 9 8 2 0 0 0 0 |
| − | 46 60 2 0 0 0 0 | + | 46 60 2 0 0 0 0 |
| − | 8 7 1 0 0 0 0 | + | 8 7 1 0 0 0 0 |
| − | 18 47 1 6 0 0 0 | + | 18 47 1 6 0 0 0 |
| − | 7 6 1 0 0 0 0 | + | 7 6 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 21 9 1 0 0 0 0 | + | 21 9 1 0 0 0 0 |
| − | 48 61 1 0 0 0 0 | + | 48 61 1 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 48 62 2 0 0 0 0 | + | 48 62 2 0 0 0 0 |
| − | 13 40 1 0 0 0 0 | + | 13 40 1 0 0 0 0 |
| − | 20 4 1 0 0 0 0 | + | 20 4 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 1 20 1 6 0 0 0 | + | 1 20 1 6 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 17 43 1 6 0 0 0 | + | 17 43 1 6 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 7 32 1 6 0 0 0 | + | 7 32 1 6 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 2 26 1 6 0 0 0 | + | 2 26 1 6 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 2 25 1 1 0 0 0 | + | 2 25 1 1 0 0 0 |
| − | 42 49 2 0 0 0 0 | + | 42 49 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 27 63 1 0 0 0 0 | + | 27 63 1 0 0 0 0 |
| − | 42 50 1 0 0 0 0 | + | 42 50 1 0 0 0 0 |
| − | 27 64 2 0 0 0 0 | + | 27 64 2 0 0 0 0 |
| − | 11 37 1 1 0 0 0 | + | 11 37 1 1 0 0 0 |
| − | 1 24 1 1 0 0 0 | + | 1 24 1 1 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPPC0007 | + | ID BMCCPPPC0007 |
| − | NAME Precorrin 6Y | + | NAME Precorrin 6Y |
| − | FORMULA C44H56N4O16 | + | FORMULA C44H56N4O16 |
| − | EXACTMASS 896.3691 | + | EXACTMASS 896.3691 |
| − | AVERAGEMASS 896.9328 | + | AVERAGEMASS 896.9328 |
| − | SMILES C(CC(O)=O)C([C@@](C)(CC(O)=O)4)=C(N=5)C[C@](C(CC(O)=O)=1)(NC(=CC([C@](CCC(O)=O)(C)2)=N[C@@H]([C@@](N3)([C@](C)(CC(O)=O)[C@H](CCC(O)=O)C3=CC45)C)[C@H](CC(O)=O)2)C1CCC(O)=O)C | + | SMILES C(CC(O)=O)C([C@@](C)(CC(O)=O)4)=C(N=5)C[C@](C(CC(O)=O)=1)(NC(=CC([C@](CCC(O)=O)(C)2)=N[C@@H]([C@@](N3)([C@](C)(CC(O)=O)[C@H](CCC(O)=O)C3=CC45)C)[C@H](CC(O)=O)2)C1CCC(O)=O)C |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06319 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06319 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 68 0 0 1 0 0 0 0 0999 V2000
7.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
6.3666 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
5.3884 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.7620 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4530 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5575 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9530 -1.7759 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
5.7620 -2.6744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
6.8666 -2.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8447 -2.3906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8228 -2.1827 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
9.9274 -2.6744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.7364 -1.7759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.1319 -0.7288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.2364 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9274 1.2167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.3010 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
9.3228 3.0773 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
8.3447 2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
6.4312 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.0575 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.6319 -1.5949 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.2582 1.9599 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.1537 2.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9543 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7788 3.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1855 4.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4373 2.6756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6942 2.0065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7431 2.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0870 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2099 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2588 -2.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5541 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6031 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3951 -4.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1537 -2.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.1353 -3.6526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.0863 -3.9616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.9044 -2.0038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3112 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.3057 -3.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.2520 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.8888 3.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.8833 3.0711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.4711 3.8801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.3228 4.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.1889 4.5773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.8935 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.7124 -3.9354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1383 -5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4441 -5.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0509 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5157 -2.8052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5352 3.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 1.6464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.8295 -3.2925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.2943 -4.9397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.0643 4.7937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.4656 3.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.0549 4.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.1889 5.5773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.1800 4.9044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5977 5.6089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17 44 1 1 0 0 0
11 12 1 0 0 0 0
35 36 1 0 0 0 0
12 13 2 0 0 0 0
36 51 1 0 0 0 0
13 14 1 0 0 0 0
36 52 2 0 0 0 0
11 22 1 6 0 0 0
32 33 1 0 0 0 0
18 19 1 0 0 0 0
33 53 1 0 0 0 0
28 29 1 0 0 0 0
33 54 2 0 0 0 0
44 45 1 0 0 0 0
29 30 1 0 0 0 0
23 16 2 0 0 0 0
30 55 1 0 0 0 0
16 17 1 0 0 0 0
30 56 2 0 0 0 0
17 18 1 0 0 0 0
19 23 1 1 0 0 0
16 15 1 0 0 0 0
15 14 2 0 0 0 0
12 38 1 0 0 0 0
45 46 1 0 0 0 0
38 39 1 0 0 0 0
11 10 1 0 0 0 0
39 57 1 0 0 0 0
10 9 1 0 0 0 0
39 58 2 0 0 0 0
6 21 2 0 0 0 0
7 31 1 1 0 0 0
3 28 1 1 0 0 0
46 59 1 0 0 0 0
9 8 2 0 0 0 0
46 60 2 0 0 0 0
8 7 1 0 0 0 0
18 47 1 6 0 0 0
7 6 1 0 0 0 0
47 48 1 0 0 0 0
21 9 1 0 0 0 0
48 61 1 0 0 0 0
2 1 1 0 0 0 0
48 62 2 0 0 0 0
13 40 1 0 0 0 0
20 4 1 0 0 0 0
4 3 1 0 0 0 0
3 2 1 0 0 0 0
1 20 1 6 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
17 43 1 6 0 0 0
1 19 1 0 0 0 0
40 41 1 0 0 0 0
7 32 1 6 0 0 0
41 42 1 0 0 0 0
8 34 1 0 0 0 0
2 26 1 6 0 0 0
34 35 1 0 0 0 0
2 25 1 1 0 0 0
42 49 2 0 0 0 0
26 27 1 0 0 0 0
14 22 1 0 0 0 0
27 63 1 0 0 0 0
42 50 1 0 0 0 0
27 64 2 0 0 0 0
11 37 1 1 0 0 0
1 24 1 1 0 0 0
S SKP 7
ID BMCCPPPC0007
NAME Precorrin 6Y
FORMULA C44H56N4O16
EXACTMASS 896.3691
AVERAGEMASS 896.9328
SMILES C(CC(O)=O)C([C@@](C)(CC(O)=O)4)=C(N=5)C[C@](C(CC(O)=O)=1)(NC(=CC([C@](CCC(O)=O)(C)2)=N[C@@H]([C@@](N3)([C@](C)(CC(O)=O)[C@H](CCC(O)=O)C3=CC45)C)[C@H](CC(O)=O)2)C1CCC(O)=O)C
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06319
M END