Mol:BMCCPPCB0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 82 92 0 0 1 0 0 0 0 0999 V2000 | + | 82 92 0 0 1 0 0 0 0 0999 V2000 |
| − | 4.2125 -4.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2125 -4.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0833 -5.0176 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 | + | 7.0833 -5.0176 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 6.3236 -5.2669 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 6.3236 -5.2669 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 7.7166 -5.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7166 -5.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3698 -4.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3698 -4.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0675 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 6.0675 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.6488 -4.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6488 -4.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4484 -4.9180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4484 -4.9180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7968 -6.2989 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7968 -6.2989 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2282 -4.1944 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2282 -4.1944 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2481 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.2481 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.9936 -5.2322 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.9936 -5.2322 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 9.1926 -5.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1926 -5.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5822 -6.6468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5822 -6.6468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9838 -6.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9838 -6.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2881 -6.1279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2881 -6.1279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8485 -4.7577 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8485 -4.7577 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4588 -6.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4588 -6.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3801 -4.9769 0.0000 Co 0 2 0 0 0 0 0 0 0 0 0 0 | + | 1.3801 -4.9769 0.0000 Co 0 2 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2645 -4.1918 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2645 -4.1918 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5313 -4.1668 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5313 -4.1668 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5291 -5.8709 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5291 -5.8709 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0453 -3.0150 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0453 -3.0150 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0328 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0328 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7165 -1.8157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7165 -1.8157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6200 -6.4674 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.6200 -6.4674 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.7962 -7.0116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7962 -7.0116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8772 -7.8020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8772 -7.8020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6046 -8.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6046 -8.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6782 -8.9240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6782 -8.9240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4099 -9.2538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4099 -9.2538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0280 -9.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0280 -9.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0555 -7.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0555 -7.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7512 -8.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7512 -8.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7548 -9.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7548 -9.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0478 -7.0256 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.0478 -7.0256 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.3797 -6.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3797 -6.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6188 -3.5728 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.6188 -3.5728 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.0381 -5.7455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0381 -5.7455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1034 -6.6300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1034 -6.6300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6568 -6.6584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6568 -6.6584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3875 -6.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3875 -6.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3542 -5.0042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3542 -5.0042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0598 -4.2693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0598 -4.2693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3771 -3.5369 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.3771 -3.5369 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.7861 -3.0119 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.7861 -3.0119 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.0965 -3.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0965 -3.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6633 -3.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6633 -3.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2574 -4.2954 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2574 -4.2954 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3875 -3.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3875 -3.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2573 -5.7545 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2573 -5.7545 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.7833 -2.2084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7833 -2.2084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1751 -3.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1751 -3.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5667 -2.8417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5667 -2.8417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3626 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3626 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7584 -2.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7584 -2.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7666 -3.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7666 -3.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4714 -1.8061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4714 -1.8061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4687 -1.0102 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4687 -1.0102 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1623 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1623 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7088 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7088 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3943 -0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3943 -0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0154 -0.6308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0154 -0.6308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1500 -6.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1500 -6.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0127 -2.8081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0127 -2.8081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4418 -9.4157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4418 -9.4157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0712 -9.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0712 -9.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4003 -6.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4003 -6.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6808 -7.3432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6808 -7.3432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4610 -7.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4610 -7.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1809 -7.9505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1809 -7.9505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7125 -7.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7125 -7.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4125 -3.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4125 -3.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8292 -2.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8292 -2.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8074 -2.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8074 -2.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5990 -2.8725 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5990 -2.8725 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4021 -2.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4021 -2.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0375 -4.5000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0375 -4.5000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3833 -2.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3833 -2.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3833 -7.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3833 -7.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6754 -7.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6754 -7.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2458 -5.8500 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2458 -5.8500 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 8 13 1 0 0 0 0 | + | 8 13 1 0 0 0 0 |
| − | 9 14 2 0 0 0 0 | + | 9 14 2 0 0 0 0 |
| − | 11 15 1 6 0 0 0 | + | 11 15 1 6 0 0 0 |
| − | 13 16 2 0 0 0 0 | + | 13 16 2 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 8 10 1 0 0 0 0 | + | 8 10 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 14 16 1 0 0 0 0 | + | 14 16 1 0 0 0 0 |
| − | 3 2 1 1 0 0 0 | + | 3 2 1 1 0 0 0 |
| − | 2 4 1 0 0 0 0 | + | 2 4 1 0 0 0 0 |
| − | 2 5 1 0 0 0 0 | + | 2 5 1 0 0 0 0 |
| − | 6 3 1 0 0 0 0 | + | 6 3 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 4 8 2 0 0 0 0 | + | 4 8 2 0 0 0 0 |
| − | 4 9 1 0 0 0 0 | + | 4 9 1 0 0 0 0 |
| − | 5 10 2 0 0 0 0 | + | 5 10 2 0 0 0 0 |
| − | 6 11 1 0 0 0 0 | + | 6 11 1 0 0 0 0 |
| − | 7 12 1 0 0 0 0 | + | 7 12 1 0 0 0 0 |
| − | 12 1 1 1 0 0 0 | + | 12 1 1 1 0 0 0 |
| − | 6 18 1 6 0 0 0 | + | 6 18 1 6 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 37 27 1 0 0 0 0 | + | 37 27 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 55 57 2 0 0 0 0 | + | 55 57 2 0 0 0 0 |
| − | 82 39 1 0 0 0 0 | + | 82 39 1 0 0 0 0 |
| − | 52 58 1 0 0 0 0 | + | 52 58 1 0 0 0 0 |
| − | 51 26 1 4 0 0 0 | + | 51 26 1 4 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 58 60 2 0 0 0 0 | + | 58 60 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 25 61 1 0 0 0 0 | + | 25 61 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 61 63 2 0 0 0 0 | + | 61 63 2 0 0 0 0 |
| − | 36 26 1 0 0 0 0 | + | 36 26 1 0 0 0 0 |
| − | 51 22 1 0 0 0 0 | + | 51 22 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 40 1 0 0 0 0 | + | 42 40 1 0 0 0 0 |
| − | 37 64 1 0 0 0 0 | + | 37 64 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 39 43 2 0 0 0 0 | + | 39 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 47 20 1 0 0 0 0 | + | 47 20 1 0 0 0 0 |
| − | 46 65 1 6 0 0 0 | + | 46 65 1 6 0 0 0 |
| − | 23 24 1 6 0 0 0 | + | 23 24 1 6 0 0 0 |
| − | 35 66 1 0 0 0 0 | + | 35 66 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 35 67 2 0 0 0 0 | + | 35 67 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 26 68 1 1 0 0 0 | + | 26 68 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 20 44 2 0 0 0 0 | + | 20 44 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 38 49 1 0 0 0 0 | + | 38 49 1 0 0 0 0 |
| − | 69 71 2 0 0 0 0 | + | 69 71 2 0 0 0 0 |
| − | 27 28 1 6 0 0 0 | + | 27 28 1 6 0 0 0 |
| − | 21 48 2 0 0 0 0 | + | 21 48 2 0 0 0 0 |
| − | 48 23 1 0 0 0 0 | + | 48 23 1 0 0 0 0 |
| − | 23 38 1 0 0 0 0 | + | 23 38 1 0 0 0 0 |
| − | 49 21 1 0 0 0 0 | + | 49 21 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 50 47 2 0 0 0 0 | + | 50 47 2 0 0 0 0 |
| − | 36 72 1 1 0 0 0 | + | 36 72 1 1 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 46 52 1 1 0 0 0 | + | 46 52 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 45 53 1 6 0 0 0 | + | 45 53 1 6 0 0 0 |
| − | 38 73 1 1 0 0 0 | + | 38 73 1 1 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 38 74 1 6 0 0 0 | + | 38 74 1 6 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 73 75 1 0 0 0 0 | + | 73 75 1 0 0 0 0 |
| − | 40 82 2 0 0 0 0 | + | 40 82 2 0 0 0 0 |
| − | 75 76 1 0 0 0 0 | + | 75 76 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 75 77 2 0 0 0 0 | + | 75 77 2 0 0 0 0 |
| − | 49 78 1 6 0 0 0 | + | 49 78 1 6 0 0 0 |
| − | 36 33 1 6 0 0 0 | + | 36 33 1 6 0 0 0 |
| − | 50 79 1 0 0 0 0 | + | 50 79 1 0 0 0 0 |
| − | 39 37 1 0 0 0 0 | + | 39 37 1 0 0 0 0 |
| − | 42 80 1 0 0 0 0 | + | 42 80 1 0 0 0 0 |
| − | 37 81 1 0 0 0 0 | + | 37 81 1 0 0 0 0 |
| − | 22 19 1 0 0 0 0 | + | 22 19 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 19 82 1 0 0 0 0 | + | 19 82 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPCB0005 | + | ID BMCCPPCB0005 |
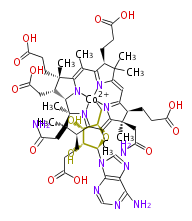
| − | NAME Adenosyl cobyrinate diamide | + | NAME Adenosyl cobyrinate diamide |
| − | FORMULA C55H73CoN11O15 | + | FORMULA C55H73CoN11O15 |
| − | EXACTMASS 1186.4619 | + | EXACTMASS 1186.4619 |
| − | AVERAGEMASS 1187.1664 | + | AVERAGEMASS 1187.1664 |
| − | SMILES O[C@H]([C@H](O)1)[C@H](n(c%11%10)cnc%10c(ncn%11)N)O[C@@H]1C[Co+2](N7=8)(N5=6)(N34)N(C2=9)=C(C(=C3[C@@]([C@@H](CC(O)=O)C([C@]5(C)[C@]([C@@H](C(C(=C7[C@@](CC(N)=O)(C)[C@H](CCC(O)=O)C(C9)8)C)6)CCC(O)=O)(C)CC(N)=O)4)(C)CCC(O)=O)C)[C@H](C2(C)C)CCC(=O)O | + | SMILES O[C@H]([C@H](O)1)[C@H](n(c%11%10)cnc%10c(ncn%11)N)O[C@@H]1C[Co+2](N7=8)(N5=6)(N34)N(C2=9)=C(C(=C3[C@@]([C@@H](CC(O)=O)C([C@]5(C)[C@]([C@@H](C(C(=C7[C@@](CC(N)=O)(C)[C@H](CCC(O)=O)C(C9)8)C)6)CCC(O)=O)(C)CC(N)=O)4)(C)CCC(O)=O)C)[C@H](C2(C)C)CCC(=O)O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06506 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06506 |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
82 92 0 0 1 0 0 0 0 0999 V2000
4.2125 -4.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0833 -5.0176 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0
6.3236 -5.2669 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
7.7166 -5.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3698 -4.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0675 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.6488 -4.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.4484 -4.9180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7968 -6.2989 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.2282 -4.1944 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.2481 -6.0097 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.9936 -5.2322 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
9.1926 -5.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5822 -6.6468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9838 -6.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.2881 -6.1279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.8485 -4.7577 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.4588 -6.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3801 -4.9769 0.0000 Co 0 2 0 0 0 0 0 0 0 0 0 0
2.2645 -4.1918 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0
0.5313 -4.1668 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0
0.5291 -5.8709 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0
-0.0453 -3.0150 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0328 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7165 -1.8157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6200 -6.4674 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.7962 -7.0116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8772 -7.8020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6046 -8.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6782 -8.9240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4099 -9.2538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0280 -9.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0555 -7.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7512 -8.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7548 -9.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0478 -7.0256 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.3797 -6.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6188 -3.5728 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.0381 -5.7455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1034 -6.6300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6568 -6.6584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3875 -6.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3542 -5.0042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0598 -4.2693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3771 -3.5369 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.7861 -3.0119 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.0965 -3.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6633 -3.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2574 -4.2954 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3875 -3.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2573 -5.7545 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.7833 -2.2084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1751 -3.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5667 -2.8417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3626 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7584 -2.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7666 -3.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4714 -1.8061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4687 -1.0102 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.1623 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7088 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3943 -0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0154 -0.6308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1500 -6.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0127 -2.8081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4418 -9.4157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0712 -9.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4003 -6.5996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6808 -7.3432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4610 -7.4712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1809 -7.9505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7125 -7.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4125 -3.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8292 -2.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8074 -2.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5990 -2.8725 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-1.4021 -2.1961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0375 -4.5000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3833 -2.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3833 -7.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6754 -7.2154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2458 -5.8500 0.0000 N 0 0 3 0 0 0 0 0 0 0 0 0
8 13 1 0 0 0 0
9 14 2 0 0 0 0
11 15 1 6 0 0 0
13 16 2 0 0 0 0
13 17 1 0 0 0 0
8 10 1 0 0 0 0
11 12 1 0 0 0 0
14 16 1 0 0 0 0
3 2 1 1 0 0 0
2 4 1 0 0 0 0
2 5 1 0 0 0 0
6 3 1 0 0 0 0
3 7 1 0 0 0 0
4 8 2 0 0 0 0
4 9 1 0 0 0 0
5 10 2 0 0 0 0
6 11 1 0 0 0 0
7 12 1 0 0 0 0
12 1 1 1 0 0 0
6 18 1 6 0 0 0
54 55 1 0 0 0 0
37 27 1 0 0 0 0
55 56 1 0 0 0 0
27 40 1 0 0 0 0
55 57 2 0 0 0 0
82 39 1 0 0 0 0
52 58 1 0 0 0 0
51 26 1 4 0 0 0
58 59 1 0 0 0 0
24 25 1 0 0 0 0
58 60 2 0 0 0 0
33 34 1 0 0 0 0
25 61 1 0 0 0 0
22 41 1 0 0 0 0
61 62 1 0 0 0 0
41 36 1 0 0 0 0
61 63 2 0 0 0 0
36 26 1 0 0 0 0
51 22 1 0 0 0 0
41 42 2 0 0 0 0
42 40 1 0 0 0 0
37 64 1 0 0 0 0
34 35 1 0 0 0 0
39 43 2 0 0 0 0
43 44 1 0 0 0 0
47 20 1 0 0 0 0
46 65 1 6 0 0 0
23 24 1 6 0 0 0
35 66 1 0 0 0 0
44 45 1 0 0 0 0
35 67 2 0 0 0 0
45 46 1 0 0 0 0
26 68 1 1 0 0 0
46 47 1 0 0 0 0
68 69 1 0 0 0 0
20 44 2 0 0 0 0
69 70 1 0 0 0 0
38 49 1 0 0 0 0
69 71 2 0 0 0 0
27 28 1 6 0 0 0
21 48 2 0 0 0 0
48 23 1 0 0 0 0
23 38 1 0 0 0 0
49 21 1 0 0 0 0
48 50 1 0 0 0 0
50 47 2 0 0 0 0
36 72 1 1 0 0 0
49 51 1 0 0 0 0
28 29 1 0 0 0 0
46 52 1 1 0 0 0
29 30 1 0 0 0 0
45 53 1 6 0 0 0
38 73 1 1 0 0 0
53 54 1 0 0 0 0
38 74 1 6 0 0 0
30 31 2 0 0 0 0
73 75 1 0 0 0 0
40 82 2 0 0 0 0
75 76 1 0 0 0 0
30 32 1 0 0 0 0
75 77 2 0 0 0 0
49 78 1 6 0 0 0
36 33 1 6 0 0 0
50 79 1 0 0 0 0
39 37 1 0 0 0 0
42 80 1 0 0 0 0
37 81 1 0 0 0 0
22 19 1 0 0 0 0
21 19 1 0 0 0 0
20 19 1 0 0 0 0
19 82 1 0 0 0 0
19 1 1 0 0 0 0
S SKP 7
ID BMCCPPCB0005
NAME Adenosyl cobyrinate diamide
FORMULA C55H73CoN11O15
EXACTMASS 1186.4619
AVERAGEMASS 1187.1664
SMILES O[C@H]([C@H](O)1)[C@H](n(c%11%10)cnc%10c(ncn%11)N)O[C@@H]1C[Co+2](N7=8)(N5=6)(N34)N(C2=9)=C(C(=C3[C@@]([C@@H](CC(O)=O)C([C@]5(C)[C@]([C@@H](C(C(=C7[C@@](CC(N)=O)(C)[C@H](CCC(O)=O)C(C9)8)C)6)CCC(O)=O)(C)CC(N)=O)4)(C)CCC(O)=O)C)[C@H](C2(C)C)CCC(=O)O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C06506
M END