Aritalab:Lecture/JSBi/Test/Bio
(New page: =タンパク質の構造= ==二次構造== タンパク質をアミノ酸の鎖として捉えたときの情報を、二次構造情報という。 二次構造には大きく分けて&...) |
|||
| Line 1: | Line 1: | ||
=タンパク質の構造= | =タンパク質の構造= | ||
| + | タンパク質を大きく分けると表のとおり。 | ||
| + | {| class="wikitable" | ||
| + | |rowspan="2"| 水溶性タンパク || 球状 (globular) || 酵素や転写因子など。分子内部は疎水性、外部は親水性。 | ||
| + | |- | ||
| + | | 不定形 (disordered) || 真核生物に多い不定構造。親水性アミノ酸が多い。とりわけEPQSRKを含む。 | ||
| + | |- | ||
| + | |colspan="2"| 膜タンパク | 受容体、トランスポーターなど。疎水性アミノ酸が多い。とりわけ膜貫通領域は疎水性。 | ||
| + | |} | ||
==二次構造== | ==二次構造== | ||
| − | + | タンパク質をアミノ酸の鎖として捉えたときの情報を、二次構造という。アミノ酸配列の先頭をN末端、末尾をC末端と呼ぶ。 | |
| − | + | 二次構造は、以下の通り。 | |
| − | * αへリックス ... | + | * αへリックス ... 3.6残基を周期として巻くらせん(へリックス) |
| − | * 平行βシート ... | + | * 平行βシート ... N→C末端の方向がそろった平行直線(ストランド) |
| − | * 逆平行βシート ... | + | * 逆平行βシート ... N→Cが互い違いの平行直線 |
| − | * ループ | + | * ループ ... へリックスやストランドを結ぶリンカー |
===ペプチド結合=== | ===ペプチド結合=== | ||
| − | + | ペプチド鎖では、各アミノ酸のカルボキシル基とアミノ基の間がペプチド結合でつながれる。 | |
| − | + | この結合は共鳴によって二重結合性を帯びるので平面構造をとる。 | |
| + | Cα(アミノ酸の中心炭素)どうしの間には炭素と窒素が1個ずつ入るが、平面が固定されるのでCα-N結合角(φ)とCα-C結合角は(ψ)の二結合ぶんで立体配置が決定できる。 | ||
| + | この二つの角度を二面角と呼ぶ。 | ||
===ラマチャンドラン マップ=== | ===ラマチャンドラン マップ=== | ||
{| | {| | ||
| | | | ||
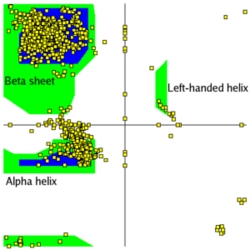
| − | ラマチャンドラン (Gopalasamudram Narayana Ramachandran) | + | ラマチャンドラン (Gopalasamudram Narayana Ramachandran) は、インド出身の生物物理学者。 |
ペプチド鎖の特徴をみるのに、各アミノ酸のφ角度とψ角度を平面にプロットする方法を考え出した。 | ペプチド鎖の特徴をみるのに、各アミノ酸のφ角度とψ角度を平面にプロットする方法を考え出した。 | ||
| − | + | こうするとへリックスとシート構造がきれいに分離され、タンパク質の構造分類に使える。 | |
| [[Image:JSBi-Ramaplot.png|250px]] | | [[Image:JSBi-Ramaplot.png|250px]] | ||
|} | |} | ||
===コンタクトマップ=== | ===コンタクトマップ=== | ||
| − | 縦( | + | 縦(上から下にN→C)横(左から右にN→C)にペプチド鎖を並べ、Cα原子間の距離が10オングストローム以内であれば色を塗る。 |
| − | + | 対角線は同一の残基がくるので黒くなる。αへリックスはこの対角線上に重なる形で表示される。 | |
| + | 平行βシートは対角線から離れた位置に同じ角度で現れ、逆平行βシートは、対角線と直行する角度で現れる。 | ||
==三次構造== | ==三次構造== | ||
| − | + | タンパク質の立体構造は三次構造とも呼ばれる。 | |
| − | === | + | 正確な立体構造はX線結晶解析やNMRで決定するしかないが、進化の観点から、構造既知の配列に似ている配列は、立体構造も似ていると仮定できる。 |
| − | + | ||
| + | {| class="wikitable" | ||
| + | |- | ||
| + | | アミノ酸の保存率 || 検出できる代表的ソフトウェア || 進化的な考察 | ||
| + | |- | ||
| + | | 30% 以上 || 殆どの配列解析ソフト || 同一祖先由来のホモロジーを持つ | ||
| + | |- | ||
| + | | 25%近辺 || Blast (E-value 10<sup>-4</sup>) || トワイライトゾーン。配列解析ソフトの限界。 | ||
| + | |- | ||
| + | | 20%以下 || PSI-Blast (E-value 10<sup>-4</sup>) || 立体構造比較が必須。 | ||
| + | |} | ||
| + | |||
| + | 三次構造比較の際には、タンパク質のドメインが保存されているか、酵素のような機能がモチーフとして保存されているかを詳細に検証する。 | ||
| + | |||
| + | ===Ab Initio構造予測=== | ||
| + | どのフォルドを取るかあらかじめ決めずに配列から構造を予測する。 | ||
| + | * 分子力学 ... エネルギーが低くなる方向に原子を移動して最適化。 | ||
| + | * 分子動力学 ... 原子をニュートンの運動方程式に従って動かして最適化。 | ||
| + | * フラグメントアセンブリ ... 9残基など決められたウィンドウ毎に既知構造ライブラリの中から形状を切り出し、ランダムに入れ替えながら構造を最適化。(ランダムに入れ替えるのでモンテカルロ法の一種。) | ||
| + | |||
| + | ===スレッディング=== | ||
| + | あらかじめ正解の構造がデータベース中にあると仮定して、類似配列を検索する。基本は配列解析。 | ||
| + | * ホモロジーモデリング ... アミノ酸が30%以上保存されるような配列を検索する。 | ||
| + | * 3D-1D法 ... タンパク質の立体構造を考える上で、20種のアミノ酸を区別する必然性は無い。各アミノ酸に環境(極性か非極性か、タンパク質の内側か外側か)分類と二次構造のカテゴリーを割り当て、翻訳されたスコアに基づいて配列相同性をみる。 | ||
===RMSD=== | ===RMSD=== | ||
| − | 三次元構造の重ね合わせ(構造アラインメント)の指標はRMSD (root mean square deviation) | + | 三次元構造の重ね合わせ(構造アラインメント)の指標はRMSD (root mean square deviation) 。 |
| + | 対応するCα原子間の距離の二乗和の平方根をとる。 | ||
Revision as of 12:56, 9 October 2010
Contents |
タンパク質の構造
タンパク質を大きく分けると表のとおり。
| 水溶性タンパク | 球状 (globular) | 酵素や転写因子など。分子内部は疎水性、外部は親水性。 |
| 不定形 (disordered) | 真核生物に多い不定構造。親水性アミノ酸が多い。とりわけEPQSRKを含む。 | |
| 膜タンパク | 受容体、トランスポーターなど。疎水性アミノ酸が多い。とりわけ膜貫通領域は疎水性。 | ||
二次構造
タンパク質をアミノ酸の鎖として捉えたときの情報を、二次構造という。アミノ酸配列の先頭をN末端、末尾をC末端と呼ぶ。 二次構造は、以下の通り。
- αへリックス ... 3.6残基を周期として巻くらせん(へリックス)
- 平行βシート ... N→C末端の方向がそろった平行直線(ストランド)
- 逆平行βシート ... N→Cが互い違いの平行直線
- ループ ... へリックスやストランドを結ぶリンカー
ペプチド結合
ペプチド鎖では、各アミノ酸のカルボキシル基とアミノ基の間がペプチド結合でつながれる。 この結合は共鳴によって二重結合性を帯びるので平面構造をとる。 Cα(アミノ酸の中心炭素)どうしの間には炭素と窒素が1個ずつ入るが、平面が固定されるのでCα-N結合角(φ)とCα-C結合角は(ψ)の二結合ぶんで立体配置が決定できる。 この二つの角度を二面角と呼ぶ。
ラマチャンドラン マップ
|
ラマチャンドラン (Gopalasamudram Narayana Ramachandran) は、インド出身の生物物理学者。 ペプチド鎖の特徴をみるのに、各アミノ酸のφ角度とψ角度を平面にプロットする方法を考え出した。 こうするとへリックスとシート構造がきれいに分離され、タンパク質の構造分類に使える。 |
|
コンタクトマップ
縦(上から下にN→C)横(左から右にN→C)にペプチド鎖を並べ、Cα原子間の距離が10オングストローム以内であれば色を塗る。 対角線は同一の残基がくるので黒くなる。αへリックスはこの対角線上に重なる形で表示される。 平行βシートは対角線から離れた位置に同じ角度で現れ、逆平行βシートは、対角線と直行する角度で現れる。
三次構造
タンパク質の立体構造は三次構造とも呼ばれる。 正確な立体構造はX線結晶解析やNMRで決定するしかないが、進化の観点から、構造既知の配列に似ている配列は、立体構造も似ていると仮定できる。
| アミノ酸の保存率 | 検出できる代表的ソフトウェア | 進化的な考察 |
| 30% 以上 | 殆どの配列解析ソフト | 同一祖先由来のホモロジーを持つ |
| 25%近辺 | Blast (E-value 10-4) | トワイライトゾーン。配列解析ソフトの限界。 |
| 20%以下 | PSI-Blast (E-value 10-4) | 立体構造比較が必須。 |
三次構造比較の際には、タンパク質のドメインが保存されているか、酵素のような機能がモチーフとして保存されているかを詳細に検証する。
Ab Initio構造予測
どのフォルドを取るかあらかじめ決めずに配列から構造を予測する。
- 分子力学 ... エネルギーが低くなる方向に原子を移動して最適化。
- 分子動力学 ... 原子をニュートンの運動方程式に従って動かして最適化。
- フラグメントアセンブリ ... 9残基など決められたウィンドウ毎に既知構造ライブラリの中から形状を切り出し、ランダムに入れ替えながら構造を最適化。(ランダムに入れ替えるのでモンテカルロ法の一種。)
スレッディング
あらかじめ正解の構造がデータベース中にあると仮定して、類似配列を検索する。基本は配列解析。
- ホモロジーモデリング ... アミノ酸が30%以上保存されるような配列を検索する。
- 3D-1D法 ... タンパク質の立体構造を考える上で、20種のアミノ酸を区別する必然性は無い。各アミノ酸に環境(極性か非極性か、タンパク質の内側か外側か)分類と二次構造のカテゴリーを割り当て、翻訳されたスコアに基づいて配列相同性をみる。
RMSD
三次元構造の重ね合わせ(構造アラインメント)の指標はRMSD (root mean square deviation) 。 対応するCα原子間の距離の二乗和の平方根をとる。