Mol:FLIAALNP0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 35 0 0 0 0 0 0 0 0999 V2000 | + | 32 35 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6210 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6210 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6210 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6210 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0647 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0647 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5084 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5084 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5084 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5084 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0647 0.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0647 0.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0479 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0479 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6042 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6042 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6042 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6042 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0479 0.3329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0479 0.3329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1603 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1603 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1603 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1603 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7551 -1.9819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7551 -1.9819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3499 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3499 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3499 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3499 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7551 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7551 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0479 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0479 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0647 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0647 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1771 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1771 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1771 0.3328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1771 0.3328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0647 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0647 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5086 1.2961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5086 1.2961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5086 1.9369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5086 1.9369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0463 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0463 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0635 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0635 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7332 0.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7332 0.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7332 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7332 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5661 -1.9815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5661 -1.9815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1786 0.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1786 0.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3013 -0.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3013 -0.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2159 -2.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2159 -2.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0820 -2.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0820 -2.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 19 2 0 0 0 0 | + | 27 19 2 0 0 0 0 |
| − | 12 28 1 0 0 0 0 | + | 12 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 34 2.5868 -1.8447 | + | M SVB 1 34 2.5868 -1.8447 |
| − | S SKP 8 | + | S SKP 8 |
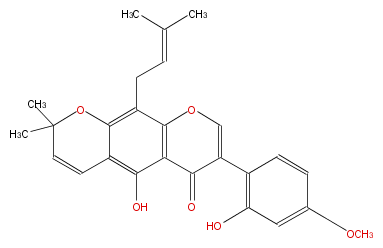
| − | ID FLIAALNP0009 | + | ID FLIAALNP0009 |
| − | KNApSAcK_ID C00009525 | + | KNApSAcK_ID C00009525 |
| − | NAME Auriculin;Auriculatin 4'-methyl ether | + | NAME Auriculin;Auriculatin 4'-methyl ether |
| − | CAS_RN 30431-68-6 | + | CAS_RN 30431-68-6 |
| − | FORMULA C26H26O6 | + | FORMULA C26H26O6 |
| − | EXACTMASS 434.172938564 | + | EXACTMASS 434.172938564 |
| − | AVERAGEMASS 434.48103999999995 | + | AVERAGEMASS 434.48103999999995 |
| − | SMILES c(c4)(c(O)cc(OC)c4)C(C3=O)=COc(c23)c(CC=C(C)C)c(c(c2O)1)OC(C=C1)(C)C | + | SMILES c(c4)(c(O)cc(OC)c4)C(C3=O)=COc(c23)c(CC=C(C)C)c(c(c2O)1)OC(C=C1)(C)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 35 0 0 0 0 0 0 0 0999 V2000
-1.6210 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6210 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0647 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5084 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5084 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0647 0.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0479 -0.9518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6042 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6042 0.0117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0479 0.3329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1603 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1603 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7551 -1.9819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3499 -1.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3499 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7551 -0.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0479 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0647 -1.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1771 -0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1771 0.3328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0647 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5086 1.2961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5086 1.9369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0463 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0635 2.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7332 0.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7332 -0.6306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5661 -1.9815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1786 0.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3013 -0.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2159 -2.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0820 -2.6385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
3 18 1 0 0 0 0
2 19 1 0 0 0 0
1 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
20 26 1 0 0 0 0
26 27 1 0 0 0 0
27 19 2 0 0 0 0
12 28 1 0 0 0 0
26 29 1 0 0 0 0
26 30 1 0 0 0 0
14 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 34
M SMT 1 OCH3
M SVB 1 34 2.5868 -1.8447
S SKP 8
ID FLIAALNP0009
KNApSAcK_ID C00009525
NAME Auriculin;Auriculatin 4'-methyl ether
CAS_RN 30431-68-6
FORMULA C26H26O6
EXACTMASS 434.172938564
AVERAGEMASS 434.48103999999995
SMILES c(c4)(c(O)cc(OC)c4)C(C3=O)=COc(c23)c(CC=C(C)C)c(c(c2O)1)OC(C=C1)(C)C
M END