Mol:FLIA1CGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1612 1.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1612 1.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3952 0.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3952 0.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9515 1.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9515 1.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5075 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5075 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5075 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5075 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0843 -0.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0843 -0.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6610 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6610 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6610 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6610 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0843 1.2379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0843 1.2379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3954 0.2628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3954 0.2628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9515 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9515 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0843 -0.7595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0843 -0.7595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2374 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2374 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2374 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2374 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7887 -1.0487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7887 -1.0487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3401 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3401 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3401 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3401 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7887 0.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7887 0.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1337 1.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1337 1.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7625 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7625 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2280 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2280 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7123 0.8257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7123 0.8257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0871 1.2006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0871 1.2006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5547 0.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5547 0.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6476 0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6476 0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3412 0.5842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3412 0.5842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9218 0.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9218 0.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9211 -1.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9211 -1.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5542 -1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5542 -1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8132 -1.1564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8132 -1.1564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2993 0.1741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2993 0.1741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9282 -0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9282 -0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3937 -0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3937 -0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8779 -0.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8779 -0.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2527 0.2725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2527 0.2725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7203 0.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7203 0.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8132 -0.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8132 -0.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5068 -0.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5068 -0.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0874 -0.6221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0874 -0.6221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1187 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1187 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3219 0.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3219 0.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9531 1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9531 1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1562 1.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1562 1.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 16 1 0 0 0 0 | + | 30 16 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 26 1 0 0 0 0 | + | 34 26 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 42 1 0 0 0 0 | + | 24 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 -8.8456 4.6326 | + | M SBV 1 45 -8.8456 4.6326 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 47 -8.8456 4.6326 | + | M SBV 2 47 -8.8456 4.6326 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIA1CGS0002 | + | ID FLIA1CGS0002 |
| − | KNApSAcK_ID C00010086 | + | KNApSAcK_ID C00010086 |
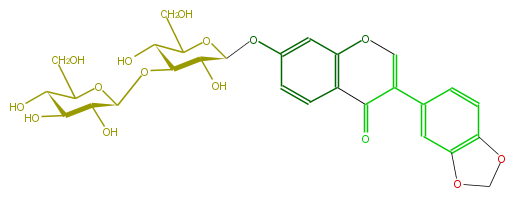
| − | NAME Pseudobaptigenin 7-O-laminaribioside | + | NAME Pseudobaptigenin 7-O-laminaribioside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C28H30O15 | + | FORMULA C28H30O15 |
| − | EXACTMASS 606.15847029 | + | EXACTMASS 606.15847029 |
| − | AVERAGEMASS 606.5288 | + | AVERAGEMASS 606.5288 |
| − | SMILES C(C(Oc(c3)ccc(C(=O)4)c3OC=C4c(c6)cc(c(c6)5)OCO5)1)(O)C(OC(O2)C(C(C(C2CO)O)O)O)C(O)C(CO)O1 | + | SMILES C(C(Oc(c3)ccc(C(=O)4)c3OC=C4c(c6)cc(c(c6)5)OCO5)1)(O)C(OC(O2)C(C(C(C2CO)O)O)O)C(O)C(CO)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-0.1612 1.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3952 0.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9515 1.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5075 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5075 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0843 -0.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6610 0.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6610 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0843 1.2379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3954 0.2628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9515 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0843 -0.7595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2374 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2374 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7887 -1.0487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3401 -0.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3401 -0.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7887 0.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1337 1.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7625 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2280 0.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7123 0.8257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0871 1.2006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5547 0.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6476 0.8056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3412 0.5842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9218 0.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9211 -1.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5542 -1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8132 -1.1564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2993 0.1741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9282 -0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3937 -0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8779 -0.1024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2527 0.2725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7203 0.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8132 -0.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5068 -0.3440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0874 -0.6221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1187 0.8099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3219 0.5963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9531 1.7380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1562 1.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
15 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 16 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 26 1 0 0 0 0
36 40 1 0 0 0 0
40 41 1 0 0 0 0
24 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 -8.8456 4.6326
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 47
M SMT 2 ^CH2OH
M SBV 2 47 -8.8456 4.6326
S SKP 8
ID FLIA1CGS0002
KNApSAcK_ID C00010086
NAME Pseudobaptigenin 7-O-laminaribioside
CAS_RN -
FORMULA C28H30O15
EXACTMASS 606.15847029
AVERAGEMASS 606.5288
SMILES C(C(Oc(c3)ccc(C(=O)4)c3OC=C4c(c6)cc(c(c6)5)OCO5)1)(O)C(OC(O2)C(C(C(C2CO)O)O)O)C(O)C(CO)O1
M END