Mol:FLIA1ACS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 43 0 0 0 0 0 0 0 0999 V2000 | + | 39 43 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7821 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7821 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7821 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7821 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0677 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0677 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6467 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6467 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6467 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6467 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0677 -0.2522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0677 -0.2522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3611 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3611 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0755 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0755 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0755 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0755 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3611 -0.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3611 -0.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3611 -2.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3611 -2.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4963 -0.2523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4963 -0.2523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7833 0.8095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7833 0.8095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0541 0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0541 0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2855 1.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2855 1.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0541 2.0128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0541 2.0128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7833 2.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7833 2.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5521 1.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5521 1.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5301 3.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5301 3.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0100 2.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0100 2.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1358 2.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1358 2.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2728 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2728 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7718 1.8892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7718 1.8892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3380 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3380 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7365 0.5585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7365 0.5585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1396 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1396 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2877 1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2877 1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7365 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7365 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 -0.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 -0.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7631 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7631 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4900 -3.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4900 -3.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2169 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2169 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2169 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2169 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4900 -1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4900 -1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7631 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7631 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9434 -3.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9434 -3.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7365 1.0437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7365 1.0437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9434 1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9434 1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 14 13 1 1 0 0 0 | + | 14 13 1 1 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 13 1 1 0 0 0 | + | 18 13 1 1 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 6 14 1 0 0 0 0 | + | 6 14 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 24 1 0 0 0 0 | + | 28 24 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 27 23 1 0 0 0 0 | + | 27 23 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 8 36 1 0 0 0 0 | + | 8 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 43 0.0000 -0.4852 | + | M SBV 1 43 0.0000 -0.4852 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FLIA1ACS0004 | + | ID FLIA1ACS0004 |
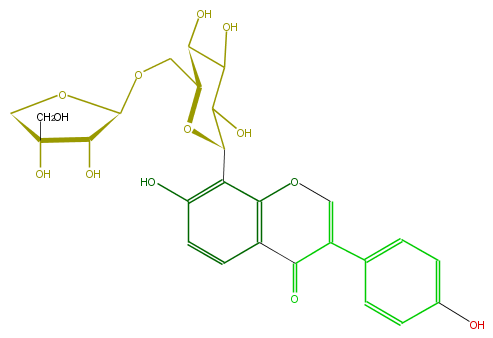
| − | FORMULA C26H28O13 | + | FORMULA C26H28O13 |
| − | EXACTMASS 548.152990982 | + | EXACTMASS 548.152990982 |
| − | AVERAGEMASS 548.49272 | + | AVERAGEMASS 548.49272 |
| − | SMILES c(c5)(c(c(c(O)c5)C(O3)C(O)C(C(C3COC(C4O)OCC4(CO)O)O)O)1)C(C(c(c2)ccc(O)c2)=CO1)=O | + | SMILES c(c5)(c(c(c(O)c5)C(O3)C(O)C(C(C3COC(C4O)OCC4(CO)O)O)O)1)C(C(c(c2)ccc(O)c2)=CO1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 43 0 0 0 0 0 0 0 0999 V2000
-0.7821 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7821 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0677 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6467 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6467 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0677 -0.2522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3611 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0755 -1.4895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0755 -0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3611 -0.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3611 -2.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4963 -0.2523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7833 0.8095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0541 0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2855 1.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0541 2.0128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7833 2.4341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5521 1.6243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5301 3.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0100 2.8516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1358 2.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2728 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7718 1.8892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3380 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7365 0.5585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1396 1.1033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2877 1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7365 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 -0.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7631 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4900 -3.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2169 -2.6890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2169 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4900 -1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7631 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9434 -3.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7365 1.0437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9434 1.4825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
14 13 1 1 0 0 0
14 15 1 0 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 1 1 0 0 0
18 13 1 1 0 0 0
17 19 1 0 0 0 0
16 20 1 0 0 0 0
18 21 1 0 0 0 0
15 22 1 0 0 0 0
6 14 1 0 0 0 0
21 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 24 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
27 23 1 0 0 0 0
31 32 2 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 31 1 0 0 0 0
8 36 1 0 0 0 0
33 37 1 0 0 0 0
38 39 1 0 0 0 0
25 38 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 CH2OH
M SBV 1 43 0.0000 -0.4852
S SKP 5
ID FLIA1ACS0004
FORMULA C26H28O13
EXACTMASS 548.152990982
AVERAGEMASS 548.49272
SMILES c(c5)(c(c(c(O)c5)C(O3)C(O)C(C(C3COC(C4O)OCC4(CO)O)O)O)1)C(C(c(c2)ccc(O)c2)=CO1)=O
M END