Mol:FL7DACGO0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4381 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4381 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4381 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4381 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1182 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1182 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6745 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6745 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6745 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6745 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1182 0.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1182 0.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2308 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2308 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7871 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7871 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7871 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7871 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2308 0.7866 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.2308 0.7866 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3432 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3432 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9102 0.4591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9102 0.4591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4772 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4772 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4772 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4772 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9102 1.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9102 1.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3432 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3432 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0440 1.7684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0440 1.7684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9942 0.7865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9942 0.7865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1182 -1.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1182 -1.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9102 2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9102 2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4675 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4675 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9519 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9519 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2094 -0.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2094 -0.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4929 -0.0098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4929 -0.0098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0135 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0135 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7720 0.2385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7720 0.2385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0440 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0440 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7557 -0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7557 -0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7839 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7839 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5187 -1.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5187 -1.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0031 -1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0031 -1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2606 -1.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2606 -1.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4620 -1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4620 -1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0648 -1.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0648 -1.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8232 -1.4527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8232 -1.4527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0952 -1.6498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0952 -1.6498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8069 -2.0367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8069 -2.0367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8352 -2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8352 -2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0675 1.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0675 1.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3531 0.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3531 0.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1188 -0.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1188 -0.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4043 -1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4043 -1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 19 1 0 0 0 0 | + | 33 19 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 43 -4.7926 5.1710 | + | M SBV 1 43 -4.7926 5.1710 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 45 -4.7926 5.1710 | + | M SBV 2 45 -4.7926 5.1710 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7DACGS0004 | + | ID FL7DACGS0004 |
| − | KNApSAcK_ID C00006626 | + | KNApSAcK_ID C00006626 |
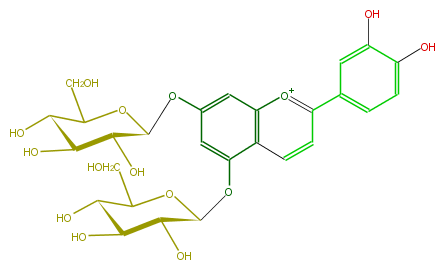
| − | NAME Luteolinidin 5,7-diglucoside | + | NAME Luteolinidin 5,7-diglucoside |
| − | CAS_RN 53947-98-1 | + | CAS_RN 53947-98-1 |
| − | FORMULA C27H31O15 | + | FORMULA C27H31O15 |
| − | EXACTMASS 595.166295322 | + | EXACTMASS 595.166295322 |
| − | AVERAGEMASS 595.52604 | + | AVERAGEMASS 595.52604 |
| − | SMILES C(C(O1)C(C(O)C(O)C1Oc(c4)cc([o+1]2)c(c4OC(O5)C(C(C(O)C5CO)O)O)ccc2c(c3)ccc(c(O)3)O)O)O | + | SMILES C(C(O1)C(C(O)C(O)C1Oc(c4)cc([o+1]2)c(c4OC(O5)C(C(C(O)C5CO)O)O)ccc2c(c3)ccc(c(O)3)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.4381 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4381 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1182 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6745 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6745 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1182 0.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2308 -0.4981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7871 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7871 0.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2308 0.7866 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.3432 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9102 0.4591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4772 0.7865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4772 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9102 1.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3432 1.4412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0440 1.7684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9942 0.7865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1182 -1.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9102 2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4675 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9519 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2094 -0.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4929 -0.0098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0135 0.5109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7720 0.2385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0440 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7557 -0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7839 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5187 -1.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0031 -1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2606 -1.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4620 -1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0648 -1.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8232 -1.4527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0952 -1.6498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8069 -2.0367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8352 -2.4230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0675 1.0460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3531 0.6335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1188 -0.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4043 -1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 19 1 0 0 0 0
26 39 1 0 0 0 0
39 40 1 0 0 0 0
35 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 ^CH2OH
M SBV 1 43 -4.7926 5.1710
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 ^CH2OH
M SBV 2 45 -4.7926 5.1710
S SKP 8
ID FL7DACGS0004
KNApSAcK_ID C00006626
NAME Luteolinidin 5,7-diglucoside
CAS_RN 53947-98-1
FORMULA C27H31O15
EXACTMASS 595.166295322
AVERAGEMASS 595.52604
SMILES C(C(O1)C(C(O)C(O)C1Oc(c4)cc([o+1]2)c(c4OC(O5)C(C(C(O)C5CO)O)O)ccc2c(c3)ccc(c(O)3)O)O)O
M END