Mol:FL7AAHGL0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 43 0 0 0 0 0 0 0 0999 V2000 | + | 40 43 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.9195 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9195 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9195 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9195 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3632 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3632 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8069 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8069 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8069 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8069 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3632 1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3632 1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2506 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2506 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6943 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6943 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6943 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6943 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2506 1.1967 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.2506 1.1967 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1382 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1382 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5712 0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5712 0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0042 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0042 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0042 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0042 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5712 2.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5712 2.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1382 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1382 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4756 1.1966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4756 1.1966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8618 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8618 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3632 -0.7301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3632 -0.7301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2782 -0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2782 -0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2247 -0.5113 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.2247 -0.5113 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0436 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0436 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.4722 -0.8285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4722 -0.8285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9912 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9912 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.2595 -0.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2595 -0.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7436 -0.6587 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.7436 -0.6587 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.1399 -0.1466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1399 -0.1466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8853 -0.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8853 -0.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5212 -0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5212 -0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3936 -1.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3936 -1.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1724 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1724 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8421 -2.9135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8421 -2.9135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3952 -3.1716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3952 -3.1716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5472 -3.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5472 -3.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5791 -4.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5791 -4.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9536 -4.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9536 -4.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5626 0.8694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5626 0.8694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2326 -4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2326 -4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2878 2.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2878 2.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1535 3.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1535 3.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 13 37 1 0 0 0 0 | + | 13 37 1 0 0 0 0 |
| − | 35 38 2 0 0 0 0 | + | 35 38 2 0 0 0 0 |
| − | 15 39 1 0 0 0 0 | + | 15 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 39 40 | + | M SAL 2 2 39 40 |
| − | M SBL 2 1 42 | + | M SBL 2 1 42 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 42 -0.2878 2.7548 | + | M SVB 2 42 -0.2878 2.7548 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 35 38 36 | + | M SAL 1 3 35 38 36 |
| − | M SBL 1 1 38 | + | M SBL 1 1 38 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 38 3.9972 -2.0326 | + | M SVB 1 38 3.9972 -2.0326 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAHGL0010 | + | ID FL7AAHGL0010 |
| − | KNApSAcK_ID C00006895 | + | KNApSAcK_ID C00006895 |
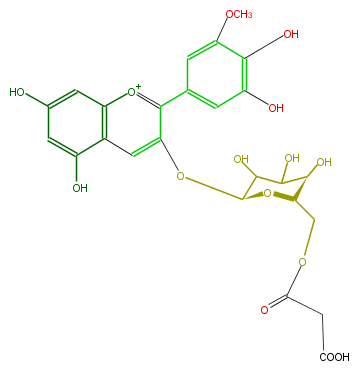
| − | NAME Petunidin 3-(6''-malonylglucoside) | + | NAME Petunidin 3-(6''-malonylglucoside) |
| − | CAS_RN 122856-10-4 | + | CAS_RN 122856-10-4 |
| − | FORMULA C25H25O15 | + | FORMULA C25H25O15 |
| − | EXACTMASS 565.11934513 | + | EXACTMASS 565.11934513 |
| − | AVERAGEMASS 565.457 | + | AVERAGEMASS 565.457 |
| − | SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c4)c([o+1]c(c34)cc(cc(O)3)O)c(c2)cc(O)c(c2OC)O)C(C(O)1)O | + | SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c4)c([o+1]c(c34)cc(cc(O)3)O)c(c2)cc(O)c(c2OC)O)C(C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 43 0 0 0 0 0 0 0 0999 V2000
-3.9195 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9195 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3632 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8069 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8069 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3632 1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2506 -0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6943 0.2332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6943 0.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2506 1.1967 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.1382 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5712 0.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0042 1.1966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0042 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5712 2.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1382 1.8513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4756 1.1966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8618 2.3513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3632 -0.7301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2782 -0.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2247 -0.5113 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0436 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.4722 -0.8285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9912 -0.9759 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.2595 -0.5113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7436 -0.6587 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.1399 -0.1466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8853 -0.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5212 -0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3936 -1.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1724 -2.2036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8421 -2.9135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3952 -3.1716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5472 -3.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5791 -4.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9536 -4.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5626 0.8694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2326 -4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2878 2.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1535 3.6521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
13 37 1 0 0 0 0
35 38 2 0 0 0 0
15 39 1 0 0 0 0
39 40 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 39 40
M SBL 2 1 42
M SMT 2 OCH3
M SVB 2 42 -0.2878 2.7548
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 35 38 36
M SBL 1 1 38
M SMT 1 COOH
M SVB 1 38 3.9972 -2.0326
S SKP 8
ID FL7AAHGL0010
KNApSAcK_ID C00006895
NAME Petunidin 3-(6''-malonylglucoside)
CAS_RN 122856-10-4
FORMULA C25H25O15
EXACTMASS 565.11934513
AVERAGEMASS 565.457
SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c4)c([o+1]c(c34)cc(cc(O)3)O)c(c2)cc(O)c(c2OC)O)C(C(O)1)O
M END