Mol:FL7AACGL0069
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 69 75 0 0 0 0 0 0 0 0999 V2000 | + | 69 75 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1887 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1887 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1887 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1887 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6324 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6324 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0761 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0761 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0761 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0761 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6324 0.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6324 0.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5198 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5198 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9635 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9635 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9635 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9635 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5198 0.9553 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.5198 0.9553 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4074 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4074 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1596 0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1596 0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7266 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7266 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7266 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7266 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1596 1.9372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1596 1.9372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4074 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4074 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7448 0.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7448 0.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2934 1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2934 1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6324 -0.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6324 -0.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5474 -0.7289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5474 -0.7289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5517 -0.4323 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.5517 -0.4323 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.9534 -0.9626 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9534 -0.9626 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5319 -0.7376 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5319 -0.7376 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.0900 -0.7316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0900 -0.7316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6845 -0.3259 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.6845 -0.3259 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.1784 -0.5930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1784 -0.5930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3272 -0.9930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3272 -0.9930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8633 -1.2940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8633 -1.2940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0812 -0.8637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0812 -0.8637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1596 2.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1596 2.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7912 -1.2457 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.7912 -1.2457 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.4200 -1.7357 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.4200 -1.7357 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.8855 -1.5278 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.8855 -1.5278 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3697 -1.5222 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3697 -1.5222 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7445 -1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7445 -1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2121 -1.3942 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.2121 -1.3942 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.3049 -0.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3049 -0.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7704 -2.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7704 -2.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5792 -2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5792 -2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5827 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5827 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2440 -0.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2440 -0.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6845 -0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6845 -0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5011 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5011 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0368 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0368 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6372 0.2462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6372 0.2462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0368 1.2326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0368 1.2326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4686 1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4686 1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4686 2.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4686 2.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0297 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0297 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0297 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0297 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4686 3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4686 3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9076 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9076 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9076 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9076 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4686 4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4686 4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3418 -3.1785 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.3418 -3.1785 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.2403 -3.0283 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2403 -3.0283 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2126 -2.4336 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2126 -2.4336 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5032 -1.9204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5032 -1.9204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1353 -2.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1353 -2.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0789 -2.7654 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.0789 -2.7654 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.3418 -3.6513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3418 -3.6513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7459 -2.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7459 -2.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0482 -4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0482 -4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6865 -2.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6865 -2.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5868 -2.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5868 -2.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5504 3.7522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5504 3.7522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0504 4.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0504 4.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5905 3.4573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5905 3.4573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3050 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3050 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 22 27 1 0 0 0 0 | + | 22 27 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 15 30 1 0 0 0 0 | + | 15 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 44 43 1 0 0 0 0 | + | 44 43 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 58 57 1 1 0 0 0 | + | 58 57 1 1 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 57 62 1 0 0 0 0 | + | 57 62 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 27 58 1 0 0 0 0 | + | 27 58 1 0 0 0 0 |
| − | 56 63 1 0 0 0 0 | + | 56 63 1 0 0 0 0 |
| − | 60 64 1 0 0 0 0 | + | 60 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 52 66 1 0 0 0 0 | + | 52 66 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 50 68 1 0 0 0 0 | + | 50 68 1 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 64 65 | + | M SAL 4 2 64 65 |
| − | M SBL 4 1 70 | + | M SBL 4 1 70 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 70 -0.6865 -2.3525 | + | M SVB 4 70 -0.6865 -2.3525 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 40 41 | + | M SAL 3 2 40 41 |
| − | M SBL 3 1 42 | + | M SBL 3 1 42 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 42 -4.677 -0.8921 | + | M SVB 3 42 -4.677 -0.8921 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 68 69 | + | M SAL 2 2 68 69 |
| − | M SBL 2 1 74 | + | M SBL 2 1 74 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 74 4.5905 3.4573 | + | M SVB 2 74 4.5905 3.4573 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 66 67 | + | M SAL 1 2 66 67 |
| − | M SBL 1 1 72 | + | M SBL 1 1 72 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 72 2.5504 3.7522 | + | M SVB 1 72 2.5504 3.7522 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0069 | + | ID FL7AACGL0069 |
| − | KNApSAcK_ID C00006845 | + | KNApSAcK_ID C00006845 |
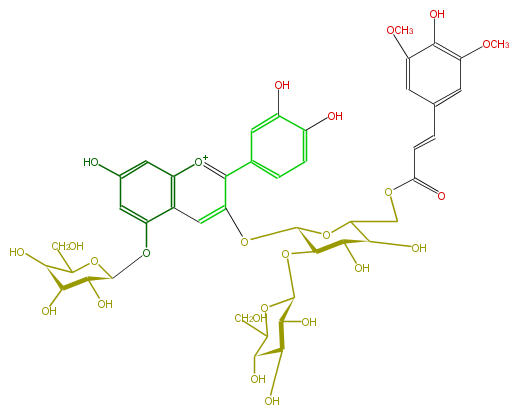
| − | NAME Cyanidin 3-(6''-sinapylsophoroside)-5-glucoside | + | NAME Cyanidin 3-(6''-sinapylsophoroside)-5-glucoside |
| − | CAS_RN 119818-02-9,107480-91-1,63003-10-1 | + | CAS_RN 119818-02-9,107480-91-1,63003-10-1 |
| − | FORMULA C44H51O25 | + | FORMULA C44H51O25 |
| − | EXACTMASS 979.271942182 | + | EXACTMASS 979.271942182 |
| − | AVERAGEMASS 979.8607400000001 | + | AVERAGEMASS 979.8607400000001 |
| − | SMILES c(c(O)1)c(c([o+1]5)c(cc(c(O[C@H](O7)[C@@H](O)[C@@H](O)[C@@H](O)C7CO)6)c5cc(O)c6)O[C@H]([C@@H]3O[C@@H]([C@H]4O)OC([C@H](O)[C@H](O)4)CO)OC([C@H]([C@H]3O)O)COC(C=Cc(c2)cc(OC)c(c(OC)2)O)=O)ccc1O | + | SMILES c(c(O)1)c(c([o+1]5)c(cc(c(O[C@H](O7)[C@@H](O)[C@@H](O)[C@@H](O)C7CO)6)c5cc(O)c6)O[C@H]([C@@H]3O[C@@H]([C@H]4O)OC([C@H](O)[C@H](O)4)CO)OC([C@H]([C@H]3O)O)COC(C=Cc(c2)cc(OC)c(c(OC)2)O)=O)ccc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
69 75 0 0 0 0 0 0 0 0999 V2000
-3.1887 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1887 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6324 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0761 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0761 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6324 0.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5198 -0.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9635 -0.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9635 0.6341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5198 0.9553 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.4074 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1596 0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7266 0.9552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7266 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1596 1.9372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4074 1.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7448 0.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2934 1.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6324 -0.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5474 -0.7289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5517 -0.4323 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.9534 -0.9626 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5319 -0.7376 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.0900 -0.7316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6845 -0.3259 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.1784 -0.5930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3272 -0.9930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8633 -1.2940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0812 -0.8637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1596 2.5917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7912 -1.2457 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.4200 -1.7357 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.8855 -1.5278 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3697 -1.5222 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7445 -1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2121 -1.3942 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.3049 -0.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7704 -2.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5792 -2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5827 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2440 -0.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6845 -0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5011 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0368 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6372 0.2462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0368 1.2326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4686 1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4686 2.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0297 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0297 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4686 3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9076 3.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9076 2.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4686 4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3418 -3.1785 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.2403 -3.0283 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2126 -2.4336 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5032 -1.9204 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1353 -2.1457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0789 -2.7654 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.3418 -3.6513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7459 -2.4263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0482 -4.1050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6865 -2.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5868 -2.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5504 3.7522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0504 4.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5905 3.4573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3050 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
22 27 1 0 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
15 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
40 41 1 0 0 0 0
34 19 1 0 0 0 0
25 42 1 0 0 0 0
42 43 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
44 43 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
55 56 1 1 0 0 0
56 57 1 1 0 0 0
58 57 1 1 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
55 61 1 0 0 0 0
57 62 1 0 0 0 0
20 21 1 0 0 0 0
27 58 1 0 0 0 0
56 63 1 0 0 0 0
60 64 1 0 0 0 0
64 65 1 0 0 0 0
52 66 1 0 0 0 0
66 67 1 0 0 0 0
50 68 1 0 0 0 0
68 69 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 64 65
M SBL 4 1 70
M SMT 4 CH2OH
M SVB 4 70 -0.6865 -2.3525
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 40 41
M SBL 3 1 42
M SMT 3 CH2OH
M SVB 3 42 -4.677 -0.8921
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 68 69
M SBL 2 1 74
M SMT 2 OCH3
M SVB 2 74 4.5905 3.4573
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 66 67
M SBL 1 1 72
M SMT 1 OCH3
M SVB 1 72 2.5504 3.7522
S SKP 8
ID FL7AACGL0069
KNApSAcK_ID C00006845
NAME Cyanidin 3-(6''-sinapylsophoroside)-5-glucoside
CAS_RN 119818-02-9,107480-91-1,63003-10-1
FORMULA C44H51O25
EXACTMASS 979.271942182
AVERAGEMASS 979.8607400000001
SMILES c(c(O)1)c(c([o+1]5)c(cc(c(O[C@H](O7)[C@@H](O)[C@@H](O)[C@@H](O)C7CO)6)c5cc(O)c6)O[C@H]([C@@H]3O[C@@H]([C@H]4O)OC([C@H](O)[C@H](O)4)CO)OC([C@H]([C@H]3O)O)COC(C=Cc(c2)cc(OC)c(c(OC)2)O)=O)ccc1O
M END