Mol:FL5FGGNS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 34 0 0 0 0 0 0 0 0999 V2000 | + | 32 34 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4388 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4388 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9950 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9950 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9951 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9951 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4387 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4387 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8825 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8825 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4388 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4388 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8824 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8824 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3262 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3262 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3262 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3262 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8725 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8725 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2299 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2299 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2299 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2299 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7969 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7969 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3639 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3639 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3638 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3638 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7969 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7969 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5511 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5511 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2472 -0.9396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2472 -0.9396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7502 -0.8678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7502 -0.8678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2299 -4.0669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2299 -4.0669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0960 -4.5669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0960 -4.5669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7969 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7969 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7969 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7969 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8824 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8824 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8824 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8824 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0815 -0.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0815 -0.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5816 0.8207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5816 0.8207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1535 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1535 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2966 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2966 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0045 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0045 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0023 -2.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0023 -2.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 6 19 1 0 0 0 0 | + | 6 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 8 25 1 0 0 0 0 | + | 8 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 2 29 1 0 0 0 0 | + | 2 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 7 SUP | + | M STY 1 7 SUP |
| − | M SLB 1 7 7 | + | M SLB 1 7 7 |
| − | M SAL 7 2 31 32 | + | M SAL 7 2 31 32 |
| − | M SBL 7 1 33 | + | M SBL 7 1 33 |
| − | M SMT 7 OCH3 | + | M SMT 7 OCH3 |
| − | M SVB 7 33 1.3907 1.7424 | + | M SVB 7 33 1.3907 1.7424 |
| − | M STY 1 6 SUP | + | M STY 1 6 SUP |
| − | M SLB 1 6 6 | + | M SLB 1 6 6 |
| − | M SAL 6 2 29 30 | + | M SAL 6 2 29 30 |
| − | M SBL 6 1 31 | + | M SBL 6 1 31 |
| − | M SMT 6 OCH3 | + | M SMT 6 OCH3 |
| − | M SVB 6 31 -2.9555 -0.6877 | + | M SVB 6 31 -2.9555 -0.6877 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 27 28 | + | M SAL 5 2 27 28 |
| − | M SBL 5 1 29 | + | M SBL 5 1 29 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 29 -2.5983 0.482 | + | M SVB 5 29 -2.5983 0.482 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 25 26 | + | M SAL 4 2 25 26 |
| − | M SBL 4 1 27 | + | M SBL 4 1 27 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 27 0.183 -0.9854 | + | M SVB 4 27 0.183 -0.9854 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 23 24 | + | M SAL 3 2 23 24 |
| − | M SBL 3 1 25 | + | M SBL 3 1 25 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 25 1.8838 -0.022 | + | M SVB 3 25 1.8838 -0.022 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 21 22 | + | M SAL 2 2 21 22 |
| − | M SBL 2 1 23 | + | M SBL 2 1 23 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 23 2.241 1.1662 | + | M SVB 2 23 2.241 1.1662 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 19 20 | + | M SAL 1 2 19 20 |
| − | M SBL 1 1 21 | + | M SBL 1 1 21 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 21 -1.4067 0.7574 | + | M SVB 1 21 -1.4067 0.7574 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FGGNS0013 | + | ID FL5FGGNS0013 |
| − | KNApSAcK_ID C00004870 | + | KNApSAcK_ID C00004870 |
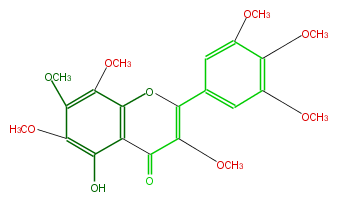
| − | NAME 5-Hydroxy-3,6,7,8,3',4',5'-heptamethoxyflavone | + | NAME 5-Hydroxy-3,6,7,8,3',4',5'-heptamethoxyflavone |
| − | CAS_RN 95165-17-6 | + | CAS_RN 95165-17-6 |
| − | FORMULA C22H24O10 | + | FORMULA C22H24O10 |
| − | EXACTMASS 448.136946988 | + | EXACTMASS 448.136946988 |
| − | AVERAGEMASS 448.41996000000006 | + | AVERAGEMASS 448.41996000000006 |
| − | SMILES O(c(c3OC)c(cc(c3)C(=C(OC)1)Oc(c2OC)c(c(O)c(c(OC)2)OC)C1=O)OC)C | + | SMILES O(c(c3OC)c(cc(c3)C(=C(OC)1)Oc(c2OC)c(c(O)c(c(OC)2)OC)C1=O)OC)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 34 0 0 0 0 0 0 0 0999 V2000
-1.4388 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9950 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9951 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4387 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8825 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4388 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8824 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3262 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3262 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8725 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2299 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2299 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7969 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3639 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3638 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7969 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5511 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2472 -0.9396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7502 -0.8678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2299 -4.0669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0960 -4.5669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7969 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7969 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8824 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8824 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0815 -0.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5816 0.8207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1535 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2966 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0045 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0023 -2.8035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
6 19 1 0 0 0 0
19 20 1 0 0 0 0
15 21 1 0 0 0 0
21 22 1 0 0 0 0
14 23 1 0 0 0 0
23 24 1 0 0 0 0
8 25 1 0 0 0 0
25 26 1 0 0 0 0
1 27 1 0 0 0 0
27 28 1 0 0 0 0
2 29 1 0 0 0 0
29 30 1 0 0 0 0
16 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 7 SUP
M SLB 1 7 7
M SAL 7 2 31 32
M SBL 7 1 33
M SMT 7 OCH3
M SVB 7 33 1.3907 1.7424
M STY 1 6 SUP
M SLB 1 6 6
M SAL 6 2 29 30
M SBL 6 1 31
M SMT 6 OCH3
M SVB 6 31 -2.9555 -0.6877
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 27 28
M SBL 5 1 29
M SMT 5 OCH3
M SVB 5 29 -2.5983 0.482
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 25 26
M SBL 4 1 27
M SMT 4 OCH3
M SVB 4 27 0.183 -0.9854
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 23 24
M SBL 3 1 25
M SMT 3 OCH3
M SVB 3 25 1.8838 -0.022
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 21 22
M SBL 2 1 23
M SMT 2 OCH3
M SVB 2 23 2.241 1.1662
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 19 20
M SBL 1 1 21
M SMT 1 OCH3
M SVB 1 21 -1.4067 0.7574
S SKP 8
ID FL5FGGNS0013
KNApSAcK_ID C00004870
NAME 5-Hydroxy-3,6,7,8,3',4',5'-heptamethoxyflavone
CAS_RN 95165-17-6
FORMULA C22H24O10
EXACTMASS 448.136946988
AVERAGEMASS 448.41996000000006
SMILES O(c(c3OC)c(cc(c3)C(=C(OC)1)Oc(c2OC)c(c(O)c(c(OC)2)OC)C1=O)OC)C
M END