Mol:FL5FGCGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.3772 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3772 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6628 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6628 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6628 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6628 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3772 4.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3772 4.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0917 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0917 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0917 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0917 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9483 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9483 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2339 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2339 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4806 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4806 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4806 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4806 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2339 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2339 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9483 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9483 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1950 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1950 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9095 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9095 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9095 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9095 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1950 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1950 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2339 1.1684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2339 1.1684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6240 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6240 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7239 1.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7239 1.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8062 4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8062 4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1950 1.1342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1950 1.1342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1950 4.2219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1950 4.2219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8088 3.1113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8088 3.1113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4448 3.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4448 3.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5722 1.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5722 1.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4828 -0.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4828 -0.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3032 -0.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3032 -0.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4338 0.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4338 0.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9615 0.8610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9615 0.8610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1410 0.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1410 0.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0103 -0.0430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0103 -0.0430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4123 -0.1580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4123 -0.1580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0299 -0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0299 -0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6118 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6118 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1973 -1.0621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1973 -1.0621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3434 0.4288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3434 0.4288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3310 -1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3310 -1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6132 -2.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6132 -2.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8712 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8712 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0487 -2.3640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0487 -2.3640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2336 -1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2336 -1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5085 -1.9496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5085 -1.9496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0068 -3.0876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0068 -3.0876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2353 -3.0843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2353 -3.0843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2930 -2.6164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2930 -2.6164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9452 -2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9452 -2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6258 -3.6197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6258 -3.6197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9080 -4.3951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9080 -4.3951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1660 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1660 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3435 -4.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3435 -4.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0612 -3.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0612 -3.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8033 -3.6871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8033 -3.6871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2887 -4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2887 -4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6333 -4.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6333 -4.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4448 -4.3525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4448 -4.3525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2398 -3.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2398 -3.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 15 25 1 0 0 0 0 | + | 15 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 28 36 1 0 0 0 0 | + | 28 36 1 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 39 44 1 0 0 0 0 | + | 39 44 1 0 0 0 0 |
| − | 38 45 1 0 0 0 0 | + | 38 45 1 0 0 0 0 |
| − | 37 46 1 0 0 0 0 | + | 37 46 1 0 0 0 0 |
| − | 41 33 1 0 0 0 0 | + | 41 33 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 1 0 0 0 | + | 48 49 1 1 0 0 0 |
| − | 50 49 1 1 0 0 0 | + | 50 49 1 1 0 0 0 |
| − | 51 50 1 1 0 0 0 | + | 51 50 1 1 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 49 54 1 0 0 0 0 | + | 49 54 1 0 0 0 0 |
| − | 48 55 1 0 0 0 0 | + | 48 55 1 0 0 0 0 |
| − | 47 56 1 0 0 0 0 | + | 47 56 1 0 0 0 0 |
| − | 51 45 1 0 0 0 0 | + | 51 45 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FGCGS0002 | + | ID FL5FGCGS0002 |
| − | FORMULA C34H42O22 | + | FORMULA C34H42O22 |
| − | EXACTMASS 802.216773028 | + | EXACTMASS 802.216773028 |
| − | AVERAGEMASS 802.68408 | + | AVERAGEMASS 802.68408 |
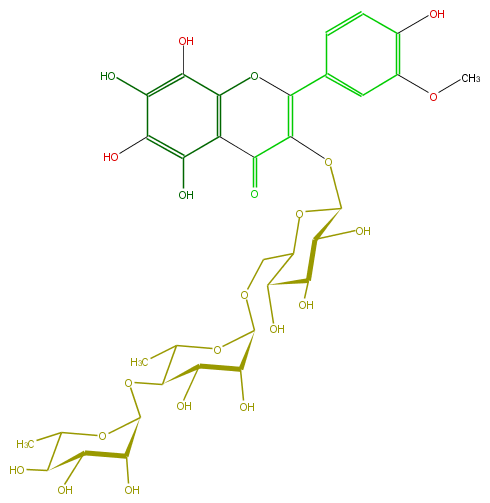
| − | SMILES c(c(OC)6)c(ccc6O)C(=C(OC(O3)C(C(C(O)C3COC(O5)C(O)C(O)C(C(C)5)OC(C4O)OC(C)C(C4O)O)O)O)2)Oc(c(O)1)c(C(=O)2)c(c(O)c1O)O | + | SMILES c(c(OC)6)c(ccc6O)C(=C(OC(O3)C(C(C(O)C3COC(O5)C(O)C(O)C(C(C)5)OC(C4O)OC(C)C(C4O)O)O)O)2)Oc(c(O)1)c(C(=O)2)c(c(O)c1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
2.3772 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6628 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6628 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3772 4.7627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0917 4.3503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0917 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9483 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2339 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4806 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4806 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2339 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9483 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1950 3.5253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9095 3.1128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9095 2.2878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1950 1.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2339 1.1684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6240 3.5253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7239 1.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8062 4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1950 1.1342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1950 4.2219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8088 3.1113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4448 3.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5722 1.9052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4828 -0.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3032 -0.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4338 0.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9615 0.8610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1410 0.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0103 -0.0430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4123 -0.1580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0299 -0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6118 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1973 -1.0621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3434 0.4288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3310 -1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6132 -2.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8712 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0487 -2.3640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2336 -1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5085 -1.9496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0068 -3.0876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2353 -3.0843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2930 -2.6164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9452 -2.1926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6258 -3.6197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9080 -4.3951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1660 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3435 -4.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0612 -3.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8033 -3.6871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2887 -4.7627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6333 -4.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4448 -4.3525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2398 -3.8313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
13 22 1 0 0 0 0
6 23 1 0 0 0 0
23 24 1 0 0 0 0
15 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
26 34 1 0 0 0 0
27 35 1 0 0 0 0
28 36 1 0 0 0 0
29 19 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 1 0 0 0
40 39 1 1 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
39 44 1 0 0 0 0
38 45 1 0 0 0 0
37 46 1 0 0 0 0
41 33 1 0 0 0 0
47 48 1 0 0 0 0
48 49 1 1 0 0 0
50 49 1 1 0 0 0
51 50 1 1 0 0 0
51 52 1 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
49 54 1 0 0 0 0
48 55 1 0 0 0 0
47 56 1 0 0 0 0
51 45 1 0 0 0 0
S SKP 5
ID FL5FGCGS0002
FORMULA C34H42O22
EXACTMASS 802.216773028
AVERAGEMASS 802.68408
SMILES c(c(OC)6)c(ccc6O)C(=C(OC(O3)C(C(C(O)C3COC(O5)C(O)C(O)C(C(C)5)OC(C4O)OC(C)C(C4O)O)O)O)2)Oc(c(O)1)c(C(=O)2)c(c(O)c1O)O
M END