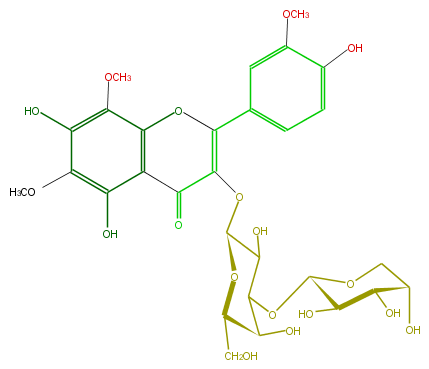
Mol:FL5FGCGA0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 51 0 0 0 0 0 0 0 0999 V2000 | + | 47 51 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5863 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5863 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5863 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5863 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8718 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8718 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1573 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1573 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1573 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1573 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8718 0.9090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8718 0.9090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4427 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4427 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2718 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2718 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2718 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2718 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4427 0.9090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4427 0.9090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4427 -1.3844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4427 -1.3844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9861 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9861 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7143 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7143 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4425 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4425 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4425 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4425 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7143 2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7143 2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9861 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9861 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3006 0.9089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3006 0.9089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0200 2.1638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0200 2.1638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7780 -0.8281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7780 -0.8281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1673 -2.0443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1673 -2.0443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5161 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5161 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6846 -2.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6846 -2.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4602 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4602 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1673 -3.6195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1673 -3.6195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9430 -2.8343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9430 -2.8343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7882 -3.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7882 -3.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8718 -1.5659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8718 -1.5659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1724 -2.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1724 -2.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7457 -3.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7457 -3.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4607 -2.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4607 -2.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1742 -2.6439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1742 -2.6439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6100 -2.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6100 -2.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9922 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9922 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7848 -3.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7848 -3.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1798 -3.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1798 -3.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4421 -3.1877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4421 -3.1877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1186 -1.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1186 -1.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1901 -3.1528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1901 -3.1528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5535 -3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5535 -3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4700 -3.7252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4700 -3.7252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2621 -0.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2621 -0.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1798 -1.2450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1798 -1.2450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7291 2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7291 2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2962 3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2962 3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8433 1.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8433 1.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2827 2.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2827 2.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 26 37 1 0 0 0 0 | + | 26 37 1 0 0 0 0 |
| − | 37 29 1 0 0 0 0 | + | 37 29 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 2 42 1 0 0 0 0 | + | 2 42 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 16 44 1 0 0 0 0 | + | 16 44 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 6 46 1 0 0 0 0 | + | 6 46 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -0.0934 0.7884 | + | M SBV 1 45 -0.0934 0.7884 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 47 0.6758 0.3864 | + | M SBV 2 47 0.6758 0.3864 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 44 45 | + | M SAL 3 2 44 45 |
| − | M SBL 3 1 49 | + | M SBL 3 1 49 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 49 -0.0149 -0.6768 | + | M SBV 3 49 -0.0149 -0.6768 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 46 47 | + | M SAL 4 2 46 47 |
| − | M SBL 4 1 51 | + | M SBL 4 1 51 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 51 -0.0285 -0.6727 | + | M SBV 4 51 -0.0285 -0.6727 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FGCGA0001 | + | ID FL5FGCGA0001 |
| − | FORMULA C29H34O18 | + | FORMULA C29H34O18 |
| − | EXACTMASS 670.174514284 | + | EXACTMASS 670.174514284 |
| − | AVERAGEMASS 670.5694599999999 | + | AVERAGEMASS 670.5694599999999 |
| − | SMILES C(C1O)(OC(C(=O)4)=C(Oc(c5OC)c(c(c(c5O)OC)O)4)c(c3)ccc(c3OC)O)OC(C(O)C(OC(O2)C(C(O)C(O)C2)O)1)CO | + | SMILES C(C1O)(OC(C(=O)4)=C(Oc(c5OC)c(c(c(c5O)OC)O)4)c(c3)ccc(c3OC)O)OC(C(O)C(OC(O2)C(C(O)C(O)C2)O)1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 51 0 0 0 0 0 0 0 0999 V2000
-2.5863 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5863 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8718 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1573 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1573 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8718 0.9090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4427 -0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2718 -0.3286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2718 0.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4427 0.9090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4427 -1.3844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9861 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7143 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4425 0.9089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4425 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7143 2.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9861 1.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3006 0.9089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0200 2.1638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7780 -0.8281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1673 -2.0443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5161 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6846 -2.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4602 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1673 -3.6195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9430 -2.8343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7882 -3.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8718 -1.5659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1724 -2.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7457 -3.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4607 -2.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1742 -2.6439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6100 -2.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9922 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7848 -3.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1798 -3.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4421 -3.1877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1186 -1.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1901 -3.1528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5535 -3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4700 -3.7252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2621 -0.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1798 -1.2450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7291 2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2962 3.9996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8433 1.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2827 2.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
25 27 1 0 0 0 0
22 20 1 0 0 0 0
3 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
31 35 1 0 0 0 0
32 36 1 0 0 0 0
26 37 1 0 0 0 0
37 29 1 0 0 0 0
21 38 1 0 0 0 0
30 39 1 0 0 0 0
40 41 1 0 0 0 0
24 40 1 0 0 0 0
42 43 1 0 0 0 0
2 42 1 0 0 0 0
44 45 1 0 0 0 0
16 44 1 0 0 0 0
46 47 1 0 0 0 0
6 46 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -0.0934 0.7884
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 47
M SMT 2 ^ OCH3
M SBV 2 47 0.6758 0.3864
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 44 45
M SBL 3 1 49
M SMT 3 OCH3
M SBV 3 49 -0.0149 -0.6768
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 46 47
M SBL 4 1 51
M SMT 4 OCH3
M SBV 4 51 -0.0285 -0.6727
S SKP 5
ID FL5FGCGA0001
FORMULA C29H34O18
EXACTMASS 670.174514284
AVERAGEMASS 670.5694599999999
SMILES C(C1O)(OC(C(=O)4)=C(Oc(c5OC)c(c(c(c5O)OC)O)4)c(c3)ccc(c3OC)O)OC(C(O)C(OC(O2)C(C(O)C(O)C2)O)1)CO
M END