Mol:FL5FGAGL0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6377 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6377 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6377 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6377 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9366 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9366 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2355 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2355 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2355 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2355 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9366 1.6740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9366 1.6740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5343 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5343 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8332 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8332 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8332 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8332 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5343 1.6740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5343 1.6740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5343 -0.7736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5343 -0.7736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1324 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1324 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5822 1.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5822 1.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2968 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2968 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2968 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2968 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5822 2.9115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5822 2.9115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1324 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1324 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9366 -0.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9366 -0.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4252 -1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4252 -1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9366 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9366 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8761 -1.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8761 -1.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8214 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8214 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3385 -1.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3385 -1.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3704 -1.5093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3704 -1.5093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6635 -1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6635 -1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2958 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2958 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8073 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8073 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7468 -0.0389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7468 -0.0389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6919 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6919 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1807 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1807 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2411 0.2704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2411 0.2704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0162 -0.0090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0162 -0.0090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8974 0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8974 0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9583 0.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9583 0.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4448 -0.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4448 -0.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1554 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1554 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3906 -2.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3906 -2.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3048 -2.5151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3048 -2.5151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8214 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8214 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5338 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5338 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3385 1.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3385 1.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0112 2.9114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0112 2.9114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9366 2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9366 2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3385 0.0551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3385 0.0551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 23 36 1 0 0 0 0 | + | 23 36 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 20 37 1 0 0 0 0 | + | 20 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 32 8 1 0 0 0 0 | + | 32 8 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 6 43 1 0 0 0 0 | + | 6 43 1 0 0 0 0 |
| − | 2 44 1 0 0 0 0 | + | 2 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FGAGL0001 | + | ID FL5FGAGL0001 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
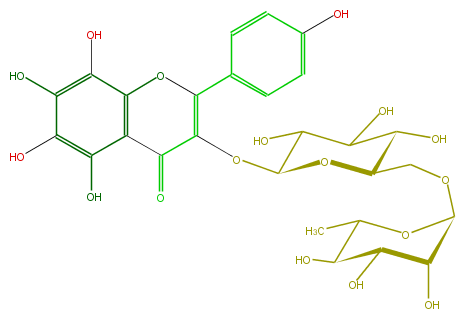
| − | SMILES C(OC(O5)C(C(O)C(O)C5C)O)C(O1)C(C(O)C(O)C1OC(=C3c(c4)ccc(O)c4)C(=O)c(c2O)c(O3)c(O)c(O)c(O)2)O | + | SMILES C(OC(O5)C(C(O)C(O)C5C)O)C(O1)C(C(O)C(O)C1OC(=C3c(c4)ccc(O)c4)C(=O)c(c2O)c(O3)c(O)c(O)c(O)2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.6377 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6377 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9366 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2355 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2355 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9366 1.6740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5343 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8332 0.4596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8332 1.2692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5343 1.6740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5343 -0.7736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1324 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5822 1.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2968 1.6739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2968 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5822 2.9115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1324 2.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9366 -0.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4252 -1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9366 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8761 -1.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8214 -2.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3385 -1.1581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3704 -1.5093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6635 -1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2958 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8073 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7468 -0.0389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6919 -0.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1807 0.5389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2411 0.2704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0162 -0.0090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8974 0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9583 0.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4448 -0.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1554 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3906 -2.0077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3048 -2.5151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8214 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5338 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3385 1.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0112 2.9114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9366 2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3385 0.0551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 33 1 0 0 0 0
30 34 1 0 0 0 0
29 35 1 0 0 0 0
23 36 1 0 0 0 0
35 36 1 0 0 0 0
20 37 1 0 0 0 0
21 38 1 0 0 0 0
22 39 1 0 0 0 0
32 8 1 0 0 0 0
27 32 1 0 0 0 0
26 40 1 0 0 0 0
1 41 1 0 0 0 0
15 42 1 0 0 0 0
6 43 1 0 0 0 0
2 44 1 0 0 0 0
S SKP 5
ID FL5FGAGL0001
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES C(OC(O5)C(C(O)C(O)C5C)O)C(O1)C(C(O)C(O)C1OC(=C3c(c4)ccc(O)c4)C(=O)c(c2O)c(O3)c(O)c(O)c(O)2)O
M END