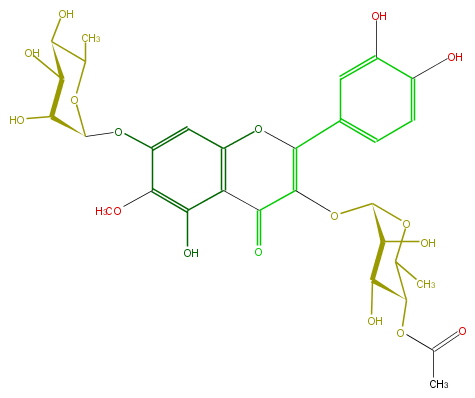
Mol:FL5FECGS0041
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 51 0 0 0 0 0 0 0 0999 V2000 | + | 47 51 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7939 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7939 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7939 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7939 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0793 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0793 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3648 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3648 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3648 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3648 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0793 1.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0793 1.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3497 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3497 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0644 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0644 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0644 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0644 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3497 1.3919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3497 1.3919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3497 -1.0597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3497 -1.0597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9640 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9640 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6923 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6923 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4206 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4206 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4206 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4206 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6923 2.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6923 2.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9640 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9640 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0793 -1.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0793 -1.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2000 2.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2000 2.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4516 1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4516 1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8590 -0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8590 -0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6064 -1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6064 -1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2941 -2.0422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2941 -2.0422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0758 -1.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0758 -1.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2941 -0.5105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2941 -0.5105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6064 -0.1135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6064 -0.1135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8246 -0.8770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8246 -0.8770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6678 -0.8770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6678 -0.8770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5989 -2.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5989 -2.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1923 -2.6718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1923 -2.6718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5705 -1.6928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5705 -1.6928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8066 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8066 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 1.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 1.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3461 1.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3461 1.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 2.7355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 2.7355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8066 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8066 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5926 2.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5926 2.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5812 3.6985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5812 3.6985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 3.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 3.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2599 2.9086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2599 2.9086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4164 1.5716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4164 1.5716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6923 3.6528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6923 3.6528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8267 -3.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8267 -3.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4164 -2.6318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4164 -2.6318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8311 -3.6985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8311 -3.6985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4697 -0.2358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4697 -0.2358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5521 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5521 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 30 43 1 0 0 0 0 | + | 30 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 2 46 1 0 0 0 0 | + | 2 46 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 46 47 | + | M SAL 1 2 46 47 |
| − | M SBL 1 1 51 | + | M SBL 1 1 51 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 51 0.6758 0.3902 | + | M SBV 1 51 0.6758 0.3902 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0041 | + | ID FL5FECGS0041 |
| − | FORMULA C30H34O17 | + | FORMULA C30H34O17 |
| − | EXACTMASS 666.179599662 | + | EXACTMASS 666.179599662 |
| − | AVERAGEMASS 666.58076 | + | AVERAGEMASS 666.58076 |
| − | SMILES O(c31)C(c(c5)ccc(O)c5O)=C(OC(O4)C(C(O)C(C4C)OC(C)=O)O)C(=O)c1c(O)c(c(c3)OC(O2)C(O)C(O)C(C2C)O)OC | + | SMILES O(c31)C(c(c5)ccc(O)c5O)=C(OC(O4)C(C(O)C(C4C)OC(C)=O)O)C(=O)c1c(O)c(c(c3)OC(O2)C(O)C(O)C(C2C)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 51 0 0 0 0 0 0 0 0999 V2000
-1.7939 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7939 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0793 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3648 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3648 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0793 1.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3497 -0.2582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0644 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0644 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3497 1.3919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3497 -1.0597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9640 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6923 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4206 1.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4206 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6923 2.8121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9640 2.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0793 -1.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2000 2.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4516 1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8590 -0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6064 -1.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2941 -2.0422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0758 -1.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2941 -0.5105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6064 -0.1135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8246 -0.8770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6678 -0.8770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5989 -2.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1923 -2.6718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5705 -1.6928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8066 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 1.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3461 1.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 2.7355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8066 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5926 2.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5812 3.6985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 3.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2599 2.9086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4164 1.5716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6923 3.6528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8267 -3.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4164 -2.6318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8311 -3.6985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4697 -0.2358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5521 -0.7657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
20 33 1 0 0 0 0
16 42 1 0 0 0 0
30 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
2 46 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 46 47
M SBL 1 1 51
M SMT 1 ^OCH3
M SBV 1 51 0.6758 0.3902
S SKP 5
ID FL5FECGS0041
FORMULA C30H34O17
EXACTMASS 666.179599662
AVERAGEMASS 666.58076
SMILES O(c31)C(c(c5)ccc(O)c5O)=C(OC(O4)C(C(O)C(C4C)OC(C)=O)O)C(=O)c1c(O)c(c(c3)OC(O2)C(O)C(O)C(C2C)O)OC
M END