Mol:FL5FECGLS001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1770 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1770 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1770 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1770 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6207 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6207 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0644 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0644 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0644 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0644 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6207 0.2218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6207 0.2218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5081 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5081 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0482 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0482 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0482 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0482 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5081 0.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5081 0.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5081 -1.5637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5081 -1.5637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6043 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6043 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1713 -0.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1713 -0.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7382 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7382 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7382 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7382 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1713 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1713 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6043 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6043 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6963 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6963 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7417 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7417 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3541 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3541 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0995 -1.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0995 -1.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8495 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8495 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2372 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2372 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4917 -1.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4917 -1.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0146 -0.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0146 -0.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7606 -0.5480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7606 -0.5480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8799 -0.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8799 -0.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3050 1.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3050 1.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6207 -1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6207 -1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0745 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0745 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3730 0.3750 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3730 0.3750 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5895 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5895 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3730 0.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3730 0.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3730 -0.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3730 -0.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1713 1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1713 1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8915 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8915 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1770 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1770 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3600 -1.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3600 -1.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0745 -1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0745 -1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 31 33 2 0 0 0 0 | + | 31 33 2 0 0 0 0 |
| − | 31 34 2 0 0 0 0 | + | 31 34 2 0 0 0 0 |
| − | 1 32 1 0 0 0 0 | + | 1 32 1 0 0 0 0 |
| − | 16 35 1 0 0 0 0 | + | 16 35 1 0 0 0 0 |
| − | 2 36 1 0 0 0 0 | + | 2 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 22 38 1 0 0 0 0 | + | 22 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 39 -6.0017 2.9208 | + | M SBV 1 39 -6.0017 2.9208 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 41 -4.7767 2.8557 | + | M SBV 2 41 -4.7767 2.8557 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECGLS001 | + | ID FL5FECGLS001 |
| − | KNApSAcK_ID C00006074 | + | KNApSAcK_ID C00006074 |
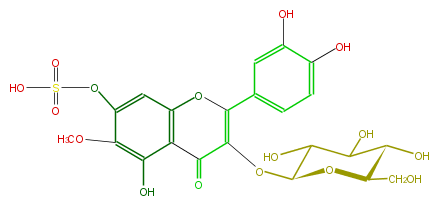
| − | NAME Patuletin 3-glucoside-7-sulfate | + | NAME Patuletin 3-glucoside-7-sulfate |
| − | CAS_RN 33530-69-7 | + | CAS_RN 33530-69-7 |
| − | FORMULA C22H22O16S | + | FORMULA C22H22O16S |
| − | EXACTMASS 574.062855346 | + | EXACTMASS 574.062855346 |
| − | AVERAGEMASS 574.46648 | + | AVERAGEMASS 574.46648 |
| − | SMILES c(O)(c13)c(OC)c(OS(O)(=O)=O)cc1OC(=C(OC(O4)C(O)C(C(C(CO)4)O)O)C3=O)c(c2)cc(c(O)c2)O | + | SMILES c(O)(c13)c(OC)c(OS(O)(=O)=O)cc1OC(=C(OC(O4)C(O)C(C(C(CO)4)O)O)C3=O)c(c2)cc(c(O)c2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-2.1770 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1770 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6207 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0644 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0644 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6207 0.2218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5081 -1.0629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0482 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0482 -0.0993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5081 0.2218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5081 -1.5637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6043 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1713 -0.1056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7382 0.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7382 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1713 1.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6043 0.8764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6963 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7417 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3541 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0995 -1.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8495 -1.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2372 -0.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4917 -1.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0146 -0.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7606 -0.5480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8799 -0.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3050 1.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6207 -1.7050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0745 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3730 0.3750 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-2.5895 0.3750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3730 0.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3730 -0.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1713 1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8915 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1770 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3600 -1.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0745 -1.8582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
31 33 2 0 0 0 0
31 34 2 0 0 0 0
1 32 1 0 0 0 0
16 35 1 0 0 0 0
2 36 1 0 0 0 0
36 37 1 0 0 0 0
22 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 39
M SMT 1 ^OCH3
M SBV 1 39 -6.0017 2.9208
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 41
M SMT 2 CH2OH
M SBV 2 41 -4.7767 2.8557
S SKP 8
ID FL5FECGLS001
KNApSAcK_ID C00006074
NAME Patuletin 3-glucoside-7-sulfate
CAS_RN 33530-69-7
FORMULA C22H22O16S
EXACTMASS 574.062855346
AVERAGEMASS 574.46648
SMILES c(O)(c13)c(OC)c(OS(O)(=O)=O)cc1OC(=C(OC(O4)C(O)C(C(C(CO)4)O)O)C3=O)c(c2)cc(c(O)c2)O
M END