Mol:FL5FECGL0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0932 -5.1801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0932 -5.1801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5394 -5.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5394 -5.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7591 -4.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7591 -4.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3462 -3.9729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3462 -3.9729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2864 -4.0844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2864 -4.0844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5061 -4.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5061 -4.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5659 -3.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5659 -3.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1530 -2.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1530 -2.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4796 -2.9886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4796 -2.9886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6993 -3.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6993 -3.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0590 -3.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0590 -3.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8924 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8924 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6685 -1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6685 -1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0893 -1.3800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0893 -1.3800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7340 -1.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7340 -1.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9579 -2.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9579 -2.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5371 -2.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5371 -2.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3128 -5.7835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3128 -5.7835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5824 -2.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5824 -2.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0330 -0.7928 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0330 -0.7928 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.6454 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.6454 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3908 -1.2512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3908 -1.2512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1408 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1408 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5285 -0.7928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5285 -0.7928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7830 -1.0058 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7830 -1.0058 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3059 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3059 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0518 -0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0518 -0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1219 -0.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1219 -0.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3913 -4.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3913 -4.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8858 -1.3752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8858 -1.3752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6002 -1.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6002 -1.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5745 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5745 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5349 -1.6511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5349 -1.6511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4480 -5.7830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4480 -5.7830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9783 -6.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9783 -6.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3768 -0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3768 -0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0196 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0196 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 2 34 1 0 0 0 0 | + | 2 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 15 36 1 0 0 0 0 | + | 15 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 30 31 | + | M SAL 4 2 30 31 |
| − | M SBL 4 1 33 | + | M SBL 4 1 33 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 33 2.8858 -1.3752 | + | M SVB 4 33 2.8858 -1.3752 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 36 37 | + | M SAL 3 2 36 37 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 39 1.5963 1.2114 | + | M SVB 3 39 1.5963 1.2114 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 34 35 | + | M SAL 2 2 34 35 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 37 -3.6002 -0.6425 | + | M SVB 2 37 -3.6002 -0.6425 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 35 0.7459 1.7877 | + | M SVB 1 35 0.7459 1.7877 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECGL0013 | + | ID FL5FECGL0013 |
| − | KNApSAcK_ID C00005680 | + | KNApSAcK_ID C00005680 |
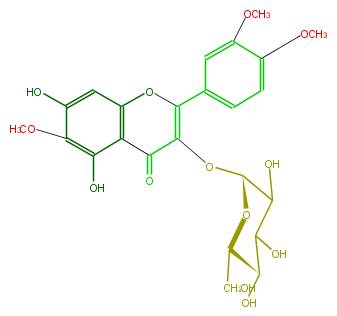
| − | NAME Quercetagetin 6,3',4'-trimethyl ether 3-glucoside | + | NAME Quercetagetin 6,3',4'-trimethyl ether 3-glucoside |
| − | CAS_RN 144599-13-3 | + | CAS_RN 144599-13-3 |
| − | FORMULA C24H26O13 | + | FORMULA C24H26O13 |
| − | EXACTMASS 522.137340918 | + | EXACTMASS 522.137340918 |
| − | AVERAGEMASS 522.45544 | + | AVERAGEMASS 522.45544 |
| − | SMILES OC([C@@H]1OC(C(=O)2)=C(c(c4)cc(OC)c(OC)c4)Oc(c3)c(c(c(OC)c(O)3)O)2)C([C@@H](O)[C@@H](CO)O1)O | + | SMILES OC([C@@H]1OC(C(=O)2)=C(c(c4)cc(OC)c(OC)c4)Oc(c3)c(c(c(OC)c(O)3)O)2)C([C@@H](O)[C@@H](CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-0.0932 -5.1801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5394 -5.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7591 -4.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3462 -3.9729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2864 -4.0844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5061 -4.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5659 -3.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1530 -2.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4796 -2.9886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6993 -3.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0590 -3.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8924 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6685 -1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0893 -1.3800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7340 -1.4937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9579 -2.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5371 -2.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3128 -5.7835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5824 -2.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0330 -0.7928 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.6454 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3908 -1.2512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1408 -1.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5285 -0.7928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7830 -1.0058 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3059 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0518 -0.5402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1219 -0.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3913 -4.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8858 -1.3752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6002 -1.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5745 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5349 -1.6511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4480 -5.7830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9783 -6.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3768 -0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0196 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
23 22 1 1 0 0 0
3 29 1 0 0 0 0
23 30 1 0 0 0 0
30 31 1 0 0 0 0
16 32 1 0 0 0 0
32 33 1 0 0 0 0
2 34 1 0 0 0 0
34 35 1 0 0 0 0
15 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 30 31
M SBL 4 1 33
M SMT 4 CH2OH
M SVB 4 33 2.8858 -1.3752
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 36 37
M SBL 3 1 39
M SMT 3 OCH3
M SVB 3 39 1.5963 1.2114
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 34 35
M SBL 2 1 37
M SMT 2 OCH3
M SVB 2 37 -3.6002 -0.6425
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 OCH3
M SVB 1 35 0.7459 1.7877
S SKP 8
ID FL5FECGL0013
KNApSAcK_ID C00005680
NAME Quercetagetin 6,3',4'-trimethyl ether 3-glucoside
CAS_RN 144599-13-3
FORMULA C24H26O13
EXACTMASS 522.137340918
AVERAGEMASS 522.45544
SMILES OC([C@@H]1OC(C(=O)2)=C(c(c4)cc(OC)c(OC)c4)Oc(c3)c(c(c(OC)c(O)3)O)2)C([C@@H](O)[C@@H](CO)O1)O
M END