Mol:FL5FDCGS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.8724 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8724 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1580 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1580 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1580 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1580 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8724 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8724 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5869 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5869 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5869 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5869 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4435 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4435 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7290 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7290 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0145 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0145 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0145 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0145 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7290 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7290 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4435 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4435 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3001 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3001 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5856 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5856 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5856 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5856 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3001 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3001 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7290 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7290 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1289 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1289 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1580 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1580 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3001 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3001 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3014 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3014 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8724 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8724 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8724 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8724 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8112 0.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8112 0.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3986 -0.3958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3986 -0.3958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6002 -0.1866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6002 -0.1866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8067 -0.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8067 -0.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2193 0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2193 0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0177 0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0177 0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1747 0.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1747 0.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6545 0.9551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6545 0.9551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2944 -0.1643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2944 -0.1643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8472 -0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8472 -0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7717 -0.8266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7717 -0.8266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8486 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8486 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4360 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4360 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6377 -0.4504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6377 -0.4504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8442 -0.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8442 -0.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2568 0.0375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2568 0.0375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0552 -0.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0552 -0.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2235 0.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2235 0.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7035 0.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7035 0.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1963 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1963 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3014 -0.4277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3014 -0.4277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4680 -1.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4680 -1.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 18 1 0 0 0 0 | + | 27 18 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 38 32 1 0 0 0 0 | + | 38 32 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FDCGS0015 | + | ID FL5FDCGS0015 |
| − | KNApSAcK_ID C00013902 | + | KNApSAcK_ID C00013902 |
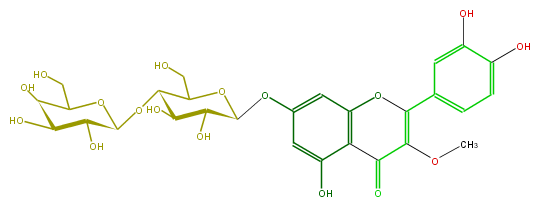
| − | NAME Quercetin 3-methyl ether 7-galactosyl-(1->4)-glucoside;2-(3,4-Dihydroxyphenyl)-7-[(4-O-beta-D-galactopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3-methyl ether 7-galactosyl-(1->4)-glucoside;2-(3,4-Dihydroxyphenyl)-7-[(4-O-beta-D-galactopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 475587-76-9 | + | CAS_RN 475587-76-9 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES c(c1C(=C5OC)Oc(c(C5=O)2)cc(OC(C(O)3)OC(CO)C(OC(O4)C(O)C(C(O)C4CO)O)C3O)cc2O)cc(c(c1)O)O | + | SMILES c(c1C(=C5OC)Oc(c(C5=O)2)cc(OC(C(O)3)OC(CO)C(OC(O4)C(O)C(C(O)C4CO)O)C3O)cc2O)cc(c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
4.8724 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1580 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1580 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8724 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5869 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5869 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4435 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7290 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0145 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0145 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7290 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4435 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3001 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5856 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5856 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3001 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7290 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1289 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1580 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3001 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3014 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8724 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8724 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8112 0.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3986 -0.3958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6002 -0.1866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8067 -0.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2193 0.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0177 0.0922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1747 0.6780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6545 0.9551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2944 -0.1643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8472 -0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7717 -0.8266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8486 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4360 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6377 -0.4504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8442 -0.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2568 0.0375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0552 -0.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2235 0.4566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7035 0.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1963 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3014 -0.4277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4680 -1.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
19 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 18 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
38 32 1 0 0 0 0
S SKP 8
ID FL5FDCGS0015
KNApSAcK_ID C00013902
NAME Quercetin 3-methyl ether 7-galactosyl-(1->4)-glucoside;2-(3,4-Dihydroxyphenyl)-7-[(4-O-beta-D-galactopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one
CAS_RN 475587-76-9
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES c(c1C(=C5OC)Oc(c(C5=O)2)cc(OC(C(O)3)OC(CO)C(OC(O4)C(O)C(C(O)C4CO)O)C3O)cc2O)cc(c(c1)O)O
M END