Mol:FL5FDCGS0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.8056 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8056 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0911 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0911 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0911 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0911 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8056 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8056 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5200 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5200 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5200 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5200 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3766 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3766 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6622 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6622 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9477 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9477 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9477 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9477 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6622 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6622 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3766 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3766 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2332 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2332 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5187 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5187 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5187 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5187 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2332 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2332 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6622 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6622 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1957 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1957 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0911 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0911 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2332 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2332 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2345 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2345 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8056 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8056 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8056 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8056 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0453 0.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0453 0.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6327 -0.6103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6327 -0.6103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8343 -0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8343 -0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0409 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0409 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4534 0.0868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4534 0.0868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2518 -0.1223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2518 -0.1223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4088 0.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4088 0.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8886 0.7406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8886 0.7406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5285 -0.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5285 -0.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0814 -0.3512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0814 -0.3512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0058 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0058 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7513 0.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7513 0.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3387 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3387 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5403 0.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5403 0.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7469 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7469 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1594 0.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1594 0.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9578 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9578 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1148 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1148 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5946 1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5946 1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2345 0.4559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2345 0.4559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7874 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7874 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7118 -0.2064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7118 -0.2064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | 27 18 1 0 0 0 0 | + | 27 18 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FDCGS0014 | + | ID FL5FDCGS0014 |
| − | KNApSAcK_ID C00013901 | + | KNApSAcK_ID C00013901 |
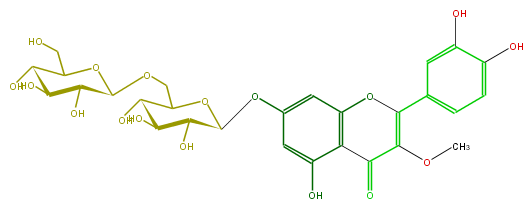
| − | NAME Madreselvin A;Quercetin 3-methyl ether 7-gentiobioside;2-(3,4-Dihydroxyphenyl)-7-[(6-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one | + | NAME Madreselvin A;Quercetin 3-methyl ether 7-gentiobioside;2-(3,4-Dihydroxyphenyl)-7-[(6-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 188682-83-9 | + | CAS_RN 188682-83-9 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES c(c1C(=C5OC)Oc(c(C5=O)4)cc(cc4O)OC(O2)C(O)C(O)C(O)C2COC(C(O)3)OC(C(O)C3O)CO)cc(c(c1)O)O | + | SMILES c(c1C(=C5OC)Oc(c(C5=O)4)cc(cc4O)OC(O2)C(O)C(O)C(O)C2COC(C(O)3)OC(C(O)C3O)CO)cc(c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
4.8056 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0911 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0911 1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8056 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5200 1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5200 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3766 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6622 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9477 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9477 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6622 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3766 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2332 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5187 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5187 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2332 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6622 -2.2687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1957 0.2062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0911 -1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2332 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.2345 1.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8056 2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8056 -1.0313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0453 0.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6327 -0.6103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8343 -0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0409 -0.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4534 0.0868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2518 -0.1223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4088 0.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8886 0.7406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5285 -0.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0814 -0.3512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0058 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7513 0.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3387 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5403 0.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7469 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1594 0.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9578 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1148 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5946 1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.2345 0.4559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7874 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7118 -0.2064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
19 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
38 31 1 0 0 0 0
27 18 1 0 0 0 0
S SKP 8
ID FL5FDCGS0014
KNApSAcK_ID C00013901
NAME Madreselvin A;Quercetin 3-methyl ether 7-gentiobioside;2-(3,4-Dihydroxyphenyl)-7-[(6-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-3-methoxy-4H-1-benzopyran-4-one
CAS_RN 188682-83-9
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES c(c1C(=C5OC)Oc(c(C5=O)4)cc(cc4O)OC(O2)C(O)C(O)C(O)C2COC(C(O)3)OC(C(O)C3O)CO)cc(c(c1)O)O
M END