Mol:FL5FAGGL0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4950 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4950 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4950 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4950 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9387 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9387 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3824 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3824 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3824 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3824 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9387 0.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9387 0.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8261 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8261 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2698 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2698 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2698 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2698 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8261 0.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8261 0.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8261 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8261 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5694 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5694 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0024 -0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0024 -0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5646 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5646 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5646 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5646 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0024 1.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0024 1.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5694 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5694 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9387 -1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9387 -1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1714 1.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1714 1.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1595 0.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1595 0.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8258 -1.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8258 -1.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0465 1.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0465 1.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1314 -0.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1314 -0.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3862 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3862 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0854 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0854 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6638 -1.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6638 -1.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2458 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2458 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5467 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5467 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9682 -1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9682 -1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1725 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1725 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2064 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2064 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3041 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3041 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2218 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2218 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9210 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9210 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4995 1.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4995 1.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0814 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0814 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3823 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3823 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8038 1.4571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8038 1.4571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6631 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6631 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0420 1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0420 1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1397 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1397 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4451 1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4451 1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1595 0.6758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1595 0.6758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6095 -1.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6095 -1.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3239 -1.8611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3239 -1.8611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 29 31 1 0 0 0 0 | + | 29 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 27 44 1 0 0 0 0 | + | 27 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -9.0879 8.5917 | + | M SBV 1 46 -9.0879 8.5917 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 44 45 | + | M SAL 2 2 44 45 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 48 -9.0879 8.5917 | + | M SBV 2 48 -9.0879 8.5917 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAGGL0006 | + | ID FL5FAGGL0006 |
| − | KNApSAcK_ID C00005745 | + | KNApSAcK_ID C00005745 |
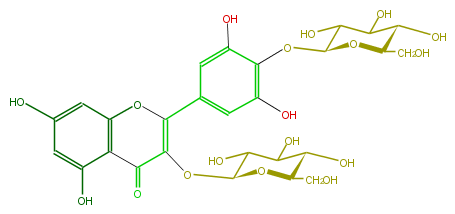
| − | NAME Myricetin 3,4'-diglucoside;3-(beta-D-Glucopyranosyloxy)-2-[4-(beta-D-glucopyranosyloxy)-3,5-dihydroxyphenyl]-5,7-dihydroxy-4H-1-benzopyran-4-one | + | NAME Myricetin 3,4'-diglucoside;3-(beta-D-Glucopyranosyloxy)-2-[4-(beta-D-glucopyranosyloxy)-3,5-dihydroxyphenyl]-5,7-dihydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 146501-37-3 | + | CAS_RN 146501-37-3 |
| − | FORMULA C27H30O18 | + | FORMULA C27H30O18 |
| − | EXACTMASS 642.143214156 | + | EXACTMASS 642.143214156 |
| − | AVERAGEMASS 642.5163 | + | AVERAGEMASS 642.5163 |
| − | SMILES C(C(C1O)OC(OC(=C(c(c5)cc(c(c5O)OC(C(O)4)OC(CO)C(O)C4O)O)3)C(=O)c(c(O3)2)c(cc(c2)O)O)C(O)C(O)1)O | + | SMILES C(C(C1O)OC(OC(=C(c(c5)cc(c(c5O)OC(C(O)4)OC(CO)C(O)C4O)O)3)C(=O)c(c(O3)2)c(cc(c2)O)O)C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-3.4950 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4950 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9387 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3824 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3824 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9387 0.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8261 -1.2275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2698 -0.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2698 -0.2640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8261 0.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8261 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5694 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0024 -0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5646 0.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5646 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0024 1.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5694 0.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9387 -1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1714 1.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1595 0.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8258 -1.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0465 1.7779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1314 -0.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3862 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0854 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6638 -1.2702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2458 -1.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5467 -0.9145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9682 -1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1725 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2064 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3041 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2218 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9210 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4995 1.2667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0814 1.1014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3823 1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8038 1.4571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6631 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0420 1.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1397 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4451 1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1595 0.6758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6095 -1.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3239 -1.8611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
14 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
24 30 1 0 0 0 0
29 31 1 0 0 0 0
28 32 1 0 0 0 0
25 21 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
38 40 1 0 0 0 0
37 41 1 0 0 0 0
34 19 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
27 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -9.0879 8.5917
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 44 45
M SBL 2 1 48
M SMT 2 CH2OH
M SBV 2 48 -9.0879 8.5917
S SKP 8
ID FL5FAGGL0006
KNApSAcK_ID C00005745
NAME Myricetin 3,4'-diglucoside;3-(beta-D-Glucopyranosyloxy)-2-[4-(beta-D-glucopyranosyloxy)-3,5-dihydroxyphenyl]-5,7-dihydroxy-4H-1-benzopyran-4-one
CAS_RN 146501-37-3
FORMULA C27H30O18
EXACTMASS 642.143214156
AVERAGEMASS 642.5163
SMILES C(C(C1O)OC(OC(=C(c(c5)cc(c(c5O)OC(C(O)4)OC(CO)C(O)C4O)O)3)C(=O)c(c(O3)2)c(cc(c2)O)O)C(O)C(O)1)O
M END