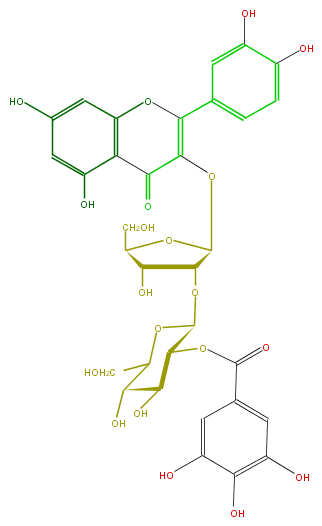
Mol:FL5FACGS0098
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.8475 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8475 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1330 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1330 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1330 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1330 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8475 4.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8475 4.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5619 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5619 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5619 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5619 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4185 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4185 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2960 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2960 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0104 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0104 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0104 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0104 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2960 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2960 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4185 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4185 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7249 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7249 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4394 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4394 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4394 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4394 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7249 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7249 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2960 1.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2960 1.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1538 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1538 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1138 1.9302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1138 1.9302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7249 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7249 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1538 4.7666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1538 4.7666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8475 5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8475 5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4361 -0.1065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4361 -0.1065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7425 -0.6480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7425 -0.6480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4361 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4361 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1158 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1158 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8014 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8014 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7478 -0.1066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7478 -0.1066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1577 0.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1577 0.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8501 -2.8488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8501 -2.8488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0253 -2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0253 -2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1651 -2.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1651 -2.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7381 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7381 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0866 -1.4518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0866 -1.4518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2771 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2771 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0437 -3.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0437 -3.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5480 -3.3430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5480 -3.3430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9075 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9075 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3515 -3.5127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3515 -3.5127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6433 -3.0896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6433 -3.0896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9227 -3.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9227 -3.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9104 -4.3162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9104 -4.3162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6186 -4.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6186 -4.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3392 -4.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3392 -4.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0465 -4.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0465 -4.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6063 -5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6063 -5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1908 -4.7175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1908 -4.7175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6556 -2.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6556 -2.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3202 -1.8821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3202 -1.8821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9606 -2.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9606 -2.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4306 -2.7313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4306 -2.7313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8014 0.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8014 0.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3129 0.8405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3129 0.8405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 27 23 1 1 0 0 0 | + | 27 23 1 1 0 0 0 |
| − | 26 28 1 1 0 0 0 | + | 26 28 1 1 0 0 0 |
| − | 28 24 1 0 0 0 0 | + | 28 24 1 0 0 0 0 |
| − | 29 26 1 0 0 0 0 | + | 29 26 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 24 1 0 0 0 0 | + | 33 24 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 42 47 1 0 0 0 0 | + | 42 47 1 0 0 0 0 |
| − | 40 48 1 0 0 0 0 | + | 40 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 38 48 1 0 0 0 0 | + | 38 48 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 35 50 1 0 0 0 0 | + | 35 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 27 52 1 0 0 0 0 | + | 27 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 56 0.6835 0.1831 | + | M SBV 1 56 0.6835 0.1831 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 58 0.0000 -0.5423 | + | M SBV 2 58 0.0000 -0.5423 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0098 | + | ID FL5FACGS0098 |
| − | FORMULA C33H32O20 | + | FORMULA C33H32O20 |
| − | EXACTMASS 748.148693464 | + | EXACTMASS 748.148693464 |
| − | AVERAGEMASS 748.59518 | + | AVERAGEMASS 748.59518 |
| − | SMILES C(O)(C1O)C(OC(c(c6)cc(c(c(O)6)O)O)=O)C(OC(C(OC(=C(c(c5)cc(c(O)c5)O)3)C(=O)c(c4O)c(cc(O)c4)O3)2)C(C(O2)CO)O)OC1CO | + | SMILES C(O)(C1O)C(OC(c(c6)cc(c(c(O)6)O)O)=O)C(OC(C(OC(=C(c(c5)cc(c(O)c5)O)3)C(=O)c(c4O)c(cc(O)c4)O3)2)C(C(O2)CO)O)OC1CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
1.8475 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1330 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1330 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8475 4.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5619 4.4248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5619 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4185 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2960 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0104 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0104 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2960 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4185 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7249 3.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4394 3.1873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4394 2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7249 1.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2960 1.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1538 3.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1138 1.9302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7249 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1538 4.7666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8475 5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4361 -0.1065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7425 -0.6480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4361 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1158 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8014 0.2535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7478 -0.1066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1577 0.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8501 -2.8488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0253 -2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1651 -2.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7381 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0866 -1.4518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2771 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0437 -3.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5480 -3.3430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9075 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3515 -3.5127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6433 -3.0896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9227 -3.4913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9104 -4.3162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6186 -4.7394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3392 -4.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0465 -4.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6063 -5.5631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1908 -4.7175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6556 -2.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3202 -1.8821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9606 -2.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4306 -2.7313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8014 0.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3129 0.8405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
28 23 1 1 0 0 0
27 23 1 1 0 0 0
26 28 1 1 0 0 0
28 24 1 0 0 0 0
29 26 1 0 0 0 0
27 29 1 0 0 0 0
23 25 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 24 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 39 1 0 0 0 0
44 45 1 0 0 0 0
43 46 1 0 0 0 0
42 47 1 0 0 0 0
40 48 1 0 0 0 0
48 49 2 0 0 0 0
38 48 1 0 0 0 0
19 26 1 0 0 0 0
50 51 1 0 0 0 0
35 50 1 0 0 0 0
52 53 1 0 0 0 0
27 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 ^ CH2OH
M SBV 1 56 0.6835 0.1831
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 CH2OH
M SBV 2 58 0.0000 -0.5423
S SKP 5
ID FL5FACGS0098
FORMULA C33H32O20
EXACTMASS 748.148693464
AVERAGEMASS 748.59518
SMILES C(O)(C1O)C(OC(c(c6)cc(c(c(O)6)O)O)=O)C(OC(C(OC(=C(c(c5)cc(c(O)c5)O)3)C(=O)c(c4O)c(cc(O)c4)O3)2)C(C(O2)CO)O)OC1CO
M END