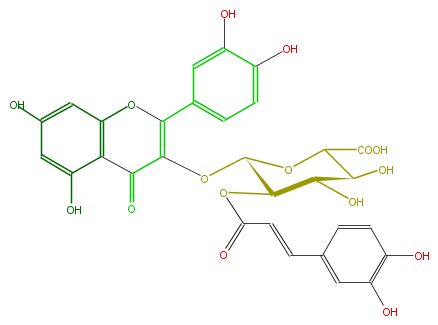
Mol:FL5FACGS0052
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5054 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5054 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5054 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5054 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8981 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8981 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2908 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2908 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2908 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2908 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8981 1.1592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8981 1.1592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6835 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6835 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0762 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0762 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0762 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0762 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6835 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6835 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6835 -0.9241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6835 -0.9241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4518 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4518 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1672 0.8317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1672 0.8317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7862 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7862 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7862 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7862 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1672 2.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1672 2.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4518 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4518 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8981 -0.9442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8981 -0.9442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4163 2.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4163 2.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0457 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0457 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2269 -0.3238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2269 -0.3238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1672 2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1672 2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5704 0.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5704 0.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1387 -0.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1387 -0.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9568 -0.3444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9568 -0.3444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7462 -0.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7462 -0.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1725 0.2380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1725 0.2380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4568 -0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4568 -0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1444 -0.6126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1444 -0.6126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7315 -0.7916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7315 -0.7916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3332 -0.1347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3332 -0.1347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5370 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5370 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1620 -1.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1620 -1.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0887 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0887 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4864 -1.8721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4864 -1.8721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1216 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1216 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4423 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4423 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0838 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0838 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4047 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4047 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0838 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0838 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4423 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4423 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0457 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0457 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4044 -2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4044 -2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9091 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9091 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4389 1.1748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4389 1.1748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4389 -0.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4389 -0.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 38 43 1 0 0 0 0 | + | 38 43 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 27 44 1 0 0 0 0 | + | 27 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 44 45 46 | + | M SAL 1 3 44 45 46 |
| − | M SBL 1 1 50 | + | M SBL 1 1 50 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SBV 1 50 -0.7366 -0.0192 | + | M SBV 1 50 -0.7366 -0.0192 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0052 | + | ID FL5FACGS0052 |
| − | FORMULA C30H24O16 | + | FORMULA C30H24O16 |
| − | EXACTMASS 640.1064347199999 | + | EXACTMASS 640.1064347199999 |
| − | AVERAGEMASS 640.50196 | + | AVERAGEMASS 640.50196 |
| − | SMILES C(OC(C(O)2)C(OC(=C4c(c5)cc(c(c5)O)O)C(c(c3O)c(O4)cc(c3)O)=O)OC(C2O)C(O)=O)(=O)C=Cc(c1)ccc(O)c(O)1 | + | SMILES C(OC(C(O)2)C(OC(=C4c(c5)cc(c(c5)O)O)C(c(c3O)c(O4)cc(c3)O)=O)OC(C2O)C(O)=O)(=O)C=Cc(c1)ccc(O)c(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-3.5054 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5054 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8981 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2908 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2908 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8981 1.1592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6835 -0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0762 0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0762 0.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6835 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6835 -0.9241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4518 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1672 0.8317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7862 1.1891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7862 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1672 2.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4518 1.9037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8981 -0.9442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4163 2.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0457 1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2269 -0.3238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1672 2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5704 0.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1387 -0.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9568 -0.3444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7462 -0.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1725 0.2380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4568 -0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1444 -0.6126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7315 -0.7916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3332 -0.1347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5370 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1620 -1.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0887 -1.2439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4864 -1.8721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1216 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4423 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0838 -2.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4047 -1.8648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0838 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4423 -1.3092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0457 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4044 -2.9756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9091 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4389 1.1748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4389 -0.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 23 1 0 0 0 0
26 31 1 0 0 0 0
29 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
38 43 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
27 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 44 45 46
M SBL 1 1 50
M SMT 1 COOH
M SBV 1 50 -0.7366 -0.0192
S SKP 5
ID FL5FACGS0052
FORMULA C30H24O16
EXACTMASS 640.1064347199999
AVERAGEMASS 640.50196
SMILES C(OC(C(O)2)C(OC(=C4c(c5)cc(c(c5)O)O)C(c(c3O)c(O4)cc(c3)O)=O)OC(C2O)C(O)=O)(=O)C=Cc(c1)ccc(O)c(O)1
M END