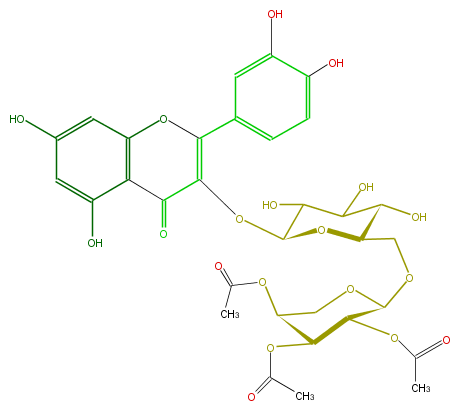
Mol:FL5FACGL0063
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 55 0 0 0 0 0 0 0 0999 V2000 | + | 51 55 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5433 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5433 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5433 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5433 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8288 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8288 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1143 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1143 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1143 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1143 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8288 1.7305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8288 1.7305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3998 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3998 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6852 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6852 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6852 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6852 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3998 1.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3998 1.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3998 -0.5628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3998 -0.5628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0290 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0290 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7573 1.3100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7573 1.3100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4855 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4855 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4855 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4855 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7573 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7573 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0290 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0290 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2576 1.7304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2576 1.7304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8288 -0.7443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8288 -0.7443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9731 2.8528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9731 2.8528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3919 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3919 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9896 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9896 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7632 -0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7632 -0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5416 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5416 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9440 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9440 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1703 -0.2284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1703 -0.2284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8004 0.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8004 0.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5891 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5891 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6362 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6362 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2201 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2201 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8768 -2.2120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8768 -2.2120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5939 -2.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5939 -2.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2772 -2.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2772 -2.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0110 -2.0630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0110 -2.0630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3328 -1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3328 -1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6287 -2.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6287 -2.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5884 -1.5354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5884 -1.5354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7369 -2.8424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7369 -2.8424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2215 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2215 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5255 -1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5255 -1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1165 -0.2728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1165 -0.2728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7573 3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7573 3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0479 -1.6230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0479 -1.6230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2932 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2932 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1916 -2.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1916 -2.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7369 -3.4674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7369 -3.4674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3132 -3.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3132 -3.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3719 -3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3719 -3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6417 -3.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6417 -3.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2576 -2.6950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2576 -2.6950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6224 -3.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6224 -3.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 38 46 1 0 0 0 0 | + | 38 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 48 2 0 0 0 0 | + | 46 48 2 0 0 0 0 |
| − | 39 49 1 0 0 0 0 | + | 39 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0063 | + | ID FL5FACGL0063 |
| − | FORMULA C32H34O19 | + | FORMULA C32H34O19 |
| − | EXACTMASS 722.1694289059999 | + | EXACTMASS 722.1694289059999 |
| − | AVERAGEMASS 722.60096 | + | AVERAGEMASS 722.60096 |
| − | SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O | + | SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 55 0 0 0 0 0 0 0 0999 V2000
-3.5433 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5433 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8288 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1143 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1143 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8288 1.7305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3998 0.0805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6852 0.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6852 1.3181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3998 1.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3998 -0.5628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0290 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7573 1.3100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4855 1.7304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4855 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7573 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0290 2.5713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2576 1.7304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8288 -0.7443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9731 2.8528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3919 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9896 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7632 -0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5416 -0.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9440 -0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1703 -0.2284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8004 0.0119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5891 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6362 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2201 -0.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8768 -2.2120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5939 -2.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2772 -2.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0110 -2.0630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3328 -1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6287 -2.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5884 -1.5354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7369 -2.8424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2215 -2.6387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5255 -1.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1165 -0.2728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7573 3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0479 -1.6230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2932 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1916 -2.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7369 -3.4674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3132 -3.8001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3719 -3.8325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6417 -3.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2576 -2.6950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6224 -3.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
38 46 1 0 0 0 0
46 47 1 0 0 0 0
46 48 2 0 0 0 0
39 49 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
S SKP 5
ID FL5FACGL0063
FORMULA C32H34O19
EXACTMASS 722.1694289059999
AVERAGEMASS 722.60096
SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O
M END