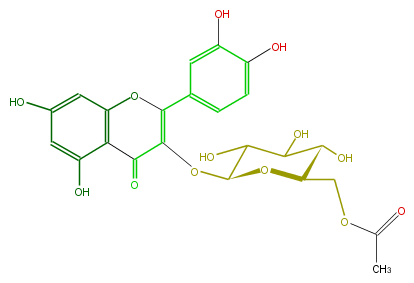
Mol:FL5FACGL0053
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1957 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1957 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1957 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1957 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6394 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6394 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0831 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0831 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0831 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0831 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6394 0.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6394 0.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5268 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5268 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9705 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9705 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9705 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9705 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5268 0.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5268 0.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5268 -0.8750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5268 -0.8750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4144 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4144 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1526 0.5831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1526 0.5831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7196 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7196 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7196 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7196 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1526 1.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1526 1.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4144 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4144 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3224 -0.6033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3224 -0.6033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7231 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7231 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3354 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3354 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0809 -0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0809 -0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8308 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8308 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2186 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2186 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4731 -0.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4731 -0.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0040 -0.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0040 -0.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7419 0.1407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7419 0.1407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6035 -0.3342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6035 -0.3342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2864 1.8923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2864 1.8923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6394 -1.0163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6394 -1.0163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8066 0.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8066 0.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4598 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4598 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6824 -1.6175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6824 -1.6175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3012 -1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3012 -1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8066 -1.4009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8066 -1.4009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3012 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3012 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1526 2.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1526 2.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 16 36 1 0 0 0 0 | + | 16 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0053 | + | ID FL5FACGL0053 |
| − | KNApSAcK_ID C00005955 | + | KNApSAcK_ID C00005955 |
| − | NAME Quercetin 3-(6''-acetylglucoside) | + | NAME Quercetin 3-(6''-acetylglucoside) |
| − | CAS_RN 54542-51-7 | + | CAS_RN 54542-51-7 |
| − | FORMULA C23H22O13 | + | FORMULA C23H22O13 |
| − | EXACTMASS 506.10604078999995 | + | EXACTMASS 506.10604078999995 |
| − | AVERAGEMASS 506.41298000000006 | + | AVERAGEMASS 506.41298000000006 |
| − | SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O | + | SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-3.1957 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1957 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6394 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0831 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0831 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6394 0.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5268 -0.3742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9705 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9705 0.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5268 0.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5268 -0.8750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4144 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1526 0.5831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7196 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7196 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1526 1.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4144 1.5651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3224 -0.6033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7231 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3354 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0809 -0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8308 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2186 -0.1119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4731 -0.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0040 -0.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7419 0.1407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6035 -0.3342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2864 1.8923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6394 -1.0163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8066 0.7878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4598 -0.7833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6824 -1.6175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3012 -1.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8066 -1.4009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3012 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1526 2.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
16 36 1 0 0 0 0
S SKP 8
ID FL5FACGL0053
KNApSAcK_ID C00005955
NAME Quercetin 3-(6''-acetylglucoside)
CAS_RN 54542-51-7
FORMULA C23H22O13
EXACTMASS 506.10604078999995
AVERAGEMASS 506.41298000000006
SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O
M END