Mol:FL5FACGL0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2749 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2749 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2749 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2749 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5605 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5605 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8459 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8459 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8459 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8459 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5605 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5605 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1314 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1314 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5831 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5831 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5831 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5831 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1314 0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1314 0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1314 -2.1862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1314 -2.1862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4827 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4827 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2110 -0.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2110 -0.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9392 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9392 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9392 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9392 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2110 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2110 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4827 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4827 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5605 -2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5605 -2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7186 1.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7186 1.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3079 -1.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3079 -1.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1284 0.1873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1284 0.1873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2110 2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2110 2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3979 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3979 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0115 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0115 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7544 -1.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7544 -1.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5020 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5020 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8884 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8884 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1454 -1.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1454 -1.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6418 -1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6418 -1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4514 -0.7260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4514 -0.7260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5297 -1.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5297 -1.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5258 0.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5258 0.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8512 0.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8512 0.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0653 0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0653 0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8512 1.5101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8512 1.5101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5258 1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5258 1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3118 1.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3118 1.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0955 2.3300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0955 2.3300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8512 1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8512 1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9172 1.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9172 1.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0943 0.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0943 0.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1766 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1766 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0943 -2.1286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0943 -2.1286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 33 21 1 0 0 0 0 | + | 33 21 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 26 42 1 0 0 0 0 | + | 26 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 47 -0.6746 -0.1140 | + | M SBV 1 47 -0.6746 -0.1140 |
| − | S SKP 5 | + | S SKP 5 |
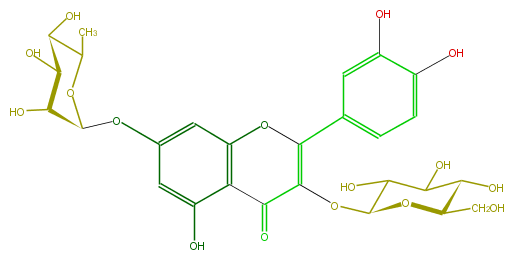
| − | ID FL5FACGL0016 | + | ID FL5FACGL0016 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(C(O)3)OC(C)C(O)C3O)cc2O | + | SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(C(O)3)OC(C)C(O)C3O)cc2O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.2749 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2749 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5605 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8459 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8459 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5605 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1314 -1.5430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5831 -1.1305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5831 -0.3054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1314 0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1314 -2.1862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4827 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2110 -0.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9392 0.2659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9392 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2110 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4827 1.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5605 -2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7186 1.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3079 -1.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1284 0.1873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2110 2.3677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3979 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0115 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7544 -1.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5020 -1.7127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8884 -1.0436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1454 -1.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6418 -1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4514 -0.7260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5297 -1.1812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5258 0.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8512 0.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0653 0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8512 1.5101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5258 1.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3118 1.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0955 2.3300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8512 1.9961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9172 1.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0943 0.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1766 -1.5988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0943 -2.1286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
24 20 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
33 21 1 0 0 0 0
42 43 1 0 0 0 0
26 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 CH2OH
M SBV 1 47 -0.6746 -0.1140
S SKP 5
ID FL5FACGL0016
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(C(O)3)OC(C)C(O)C3O)cc2O
M END