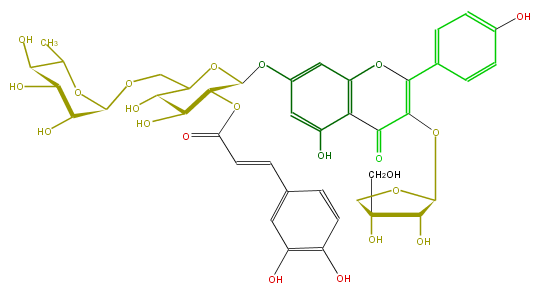
Mol:FL5FAAGS0134
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 69 0 0 0 0 0 0 0 0999 V2000 | + | 63 69 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.1025 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1025 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1025 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1025 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8170 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8170 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5314 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5314 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5314 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5314 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8170 3.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8170 3.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3880 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3880 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6736 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6736 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9591 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9591 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9591 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9591 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6736 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6736 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3880 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3880 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2446 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2446 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5301 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5301 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5301 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5301 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2446 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2446 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6736 -0.2855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6736 -0.2855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1843 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1843 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0657 0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0657 0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1322 3.1896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1322 3.1896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2446 -0.2243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2446 -0.2243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7815 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7815 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0819 0.8009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0819 0.8009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4749 1.3601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4749 1.3601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6676 1.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6676 1.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3673 1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3673 1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9743 1.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9743 1.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6875 1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6875 1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4506 1.5505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4506 1.5505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2926 0.9436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2926 0.9436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0066 0.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0066 0.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8106 0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8106 0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2513 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2513 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0492 0.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0492 0.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8223 -0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8223 -0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0013 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0013 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4303 -1.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4303 -1.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0351 -1.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0351 -1.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4648 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4648 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2895 -2.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2895 -2.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6846 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6846 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2551 -1.0985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2551 -1.0985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7185 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7185 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0558 -3.2552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0558 -3.2552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0741 2.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0741 2.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8848 1.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8848 1.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1945 1.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1945 1.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8311 0.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8311 0.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0204 0.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0204 0.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7107 1.3297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7107 1.3297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4557 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4557 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1322 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1322 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0779 2.6365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0779 2.6365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5602 2.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5602 2.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4972 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4972 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4972 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4972 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6812 -2.3272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6812 -2.3272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0491 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0491 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1319 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1319 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6812 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6812 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0911 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0911 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4972 -0.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4972 -0.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9972 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9972 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 39 44 1 0 0 0 0 | + | 39 44 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 46 53 1 0 0 0 0 | + | 46 53 1 0 0 0 0 |
| − | 45 54 1 0 0 0 0 | + | 45 54 1 0 0 0 0 |
| − | 49 29 1 0 0 0 0 | + | 49 29 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 60 55 1 1 0 0 0 | + | 60 55 1 1 0 0 0 |
| − | 59 55 1 1 0 0 0 | + | 59 55 1 1 0 0 0 |
| − | 58 60 1 1 0 0 0 | + | 58 60 1 1 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 60 57 1 0 0 0 0 | + | 60 57 1 0 0 0 0 |
| − | 61 58 1 0 0 0 0 | + | 61 58 1 0 0 0 0 |
| − | 59 61 1 0 0 0 0 | + | 59 61 1 0 0 0 0 |
| − | 19 58 1 0 0 0 0 | + | 19 58 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 55 62 1 0 0 0 0 | + | 55 62 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 69 | + | M SBL 1 1 69 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 69 0.0000 -1.0176 | + | M SBV 1 69 0.0000 -1.0176 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0134 | + | ID FL5FAAGS0134 |
| − | FORMULA C41H44O22 | + | FORMULA C41H44O22 |
| − | EXACTMASS 888.232423092 | + | EXACTMASS 888.232423092 |
| − | AVERAGEMASS 888.77486 | + | AVERAGEMASS 888.77486 |
| − | SMILES c(c13)(O)cc(OC(C(OC(=O)C=Cc(c7)cc(c(c7)O)O)5)OC(COC(C(O)6)OC(C(O)C6O)C)C(O)C5O)cc1OC(=C(OC(O4)C(C(O)(C4)CO)O)C3=O)c(c2)ccc(O)c2 | + | SMILES c(c13)(O)cc(OC(C(OC(=O)C=Cc(c7)cc(c(c7)O)O)5)OC(COC(C(O)6)OC(C(O)C6O)C)C(O)C5O)cc1OC(=C(OC(O4)C(C(O)(C4)CO)O)C3=O)c(c2)ccc(O)c2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 69 0 0 0 0 0 0 0 0999 V2000
4.1025 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1025 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8170 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5314 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5314 2.8427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8170 3.2552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3880 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6736 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9591 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9591 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6736 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3880 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2446 2.0177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5301 1.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5301 0.7802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2446 0.3677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6736 -0.2855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1843 2.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0657 0.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1322 3.1896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2446 -0.2243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7815 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0819 0.8009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4749 1.3601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6676 1.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3673 1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9743 1.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6875 1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4506 1.5505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2926 0.9436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0066 0.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8106 0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2513 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0492 0.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8223 -0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0013 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4303 -1.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0351 -1.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4648 -2.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2895 -2.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6846 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2551 -1.0985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7185 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0558 -3.2552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0741 2.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8848 1.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1945 1.4635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8311 0.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0204 0.8773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7107 1.3297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4557 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1322 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0779 2.6365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5602 2.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4972 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4972 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6812 -2.3272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0491 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1319 -1.3346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6812 -1.6946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0911 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4972 -0.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9972 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
39 44 1 0 0 0 0
32 33 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
47 52 1 0 0 0 0
46 53 1 0 0 0 0
45 54 1 0 0 0 0
49 29 1 0 0 0 0
18 25 1 0 0 0 0
60 55 1 1 0 0 0
59 55 1 1 0 0 0
58 60 1 1 0 0 0
55 56 1 0 0 0 0
60 57 1 0 0 0 0
61 58 1 0 0 0 0
59 61 1 0 0 0 0
19 58 1 0 0 0 0
62 63 1 0 0 0 0
55 62 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 69
M SMT 1 CH2OH
M SBV 1 69 0.0000 -1.0176
S SKP 5
ID FL5FAAGS0134
FORMULA C41H44O22
EXACTMASS 888.232423092
AVERAGEMASS 888.77486
SMILES c(c13)(O)cc(OC(C(OC(=O)C=Cc(c7)cc(c(c7)O)O)5)OC(COC(C(O)6)OC(C(O)C6O)C)C(O)C5O)cc1OC(=C(OC(O4)C(C(O)(C4)CO)O)C3=O)c(c2)ccc(O)c2
M END