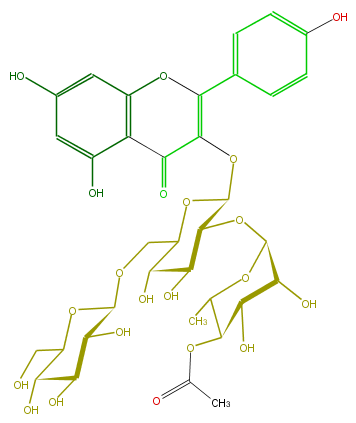
Mol:FL5FAAGS0128
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1607 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1607 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1607 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1607 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8752 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8752 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5896 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5896 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5896 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5896 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8752 3.9611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8752 3.9611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4463 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4463 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2682 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2682 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9827 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9827 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9827 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9827 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2682 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2682 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4463 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4463 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6970 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6970 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4115 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4115 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4115 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4115 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6970 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6970 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2682 0.3555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2682 0.3555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1260 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1260 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1239 1.0514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1239 1.0514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1903 3.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1903 3.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6970 0.3742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6970 0.3742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5603 -1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5603 -1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2643 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2643 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4522 -0.3659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4522 -0.3659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0234 0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0234 0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1988 0.1987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1988 0.1987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0107 -0.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0107 -0.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5792 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5792 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1470 -1.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1470 -1.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6846 -1.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6846 -1.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1935 -1.6736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1935 -1.6736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2501 -0.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2501 -0.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8481 -3.3687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8481 -3.3687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0234 -3.3376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0234 -3.3376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8356 -2.5340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8356 -2.5340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2644 -1.9384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2644 -1.9384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0891 -1.9694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0891 -1.9694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2770 -2.7730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2770 -2.7730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8257 -2.7839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8257 -2.7839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1903 -3.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1903 -3.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9958 -3.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9958 -3.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4813 -3.8003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4813 -3.8003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1422 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1422 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6664 -1.7395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6664 -1.7395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8801 -2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8801 -2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2808 -1.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2808 -1.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9932 -1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9932 -1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7798 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7798 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3789 -1.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3789 -1.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5679 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5679 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3347 -2.7218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3347 -2.7218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2621 -2.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2621 -2.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2968 -2.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2968 -2.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2479 -3.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2479 -3.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4030 -3.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4030 -3.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7549 -3.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7549 -3.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 44 53 1 0 0 0 0 | + | 44 53 1 0 0 0 0 |
| − | 52 54 1 0 0 0 0 | + | 52 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 36 29 1 0 0 0 0 | + | 36 29 1 0 0 0 0 |
| − | 48 32 1 0 0 0 0 | + | 48 32 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0128 | + | ID FL5FAAGS0128 |
| − | FORMULA C35H42O21 | + | FORMULA C35H42O21 |
| − | EXACTMASS 798.2218584059999 | + | EXACTMASS 798.2218584059999 |
| − | AVERAGEMASS 798.69538 | + | AVERAGEMASS 798.69538 |
| − | SMILES C(O5)(C(C(C(O)C5COC(O6)C(O)C(C(O)C6CO)O)O)OC(C(O)4)OC(C(C4O)OC(C)=O)C)OC(C(=O)2)=C(Oc(c3)c(c(cc3O)O)2)c(c1)ccc(O)c1 | + | SMILES C(O5)(C(C(C(O)C5COC(O6)C(O)C(C(O)C6CO)O)O)OC(C(O)4)OC(C(C4O)OC(C)=O)C)OC(C(=O)2)=C(Oc(c3)c(c(cc3O)O)2)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
1.1607 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1607 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8752 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5896 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5896 3.5486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8752 3.9611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4463 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2682 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9827 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9827 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2682 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4463 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6970 2.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4115 2.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4115 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6970 1.0737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2682 0.3555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1260 2.7236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1239 1.0514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1903 3.8954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6970 0.3742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5603 -1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2643 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4522 -0.3659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0234 0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1988 0.1987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0107 -0.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5792 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1470 -1.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6846 -1.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1935 -1.6736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2501 -0.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8481 -3.3687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0234 -3.3376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8356 -2.5340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2644 -1.9384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0891 -1.9694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2770 -2.7730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8257 -2.7839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1903 -3.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9958 -3.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4813 -3.8003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1422 -2.4083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6664 -1.7395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8801 -2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2808 -1.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9932 -1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7798 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3789 -1.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5679 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3347 -2.7218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2621 -2.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2968 -2.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2479 -3.4072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4030 -3.7718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7549 -3.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
25 19 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
46 51 1 0 0 0 0
45 52 1 0 0 0 0
44 53 1 0 0 0 0
52 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
36 29 1 0 0 0 0
48 32 1 0 0 0 0
S SKP 5
ID FL5FAAGS0128
FORMULA C35H42O21
EXACTMASS 798.2218584059999
AVERAGEMASS 798.69538
SMILES C(O5)(C(C(C(O)C5COC(O6)C(O)C(C(O)C6CO)O)O)OC(C(O)4)OC(C(C4O)OC(C)=O)C)OC(C(=O)2)=C(Oc(c3)c(c(cc3O)O)2)c(c1)ccc(O)c1
M END