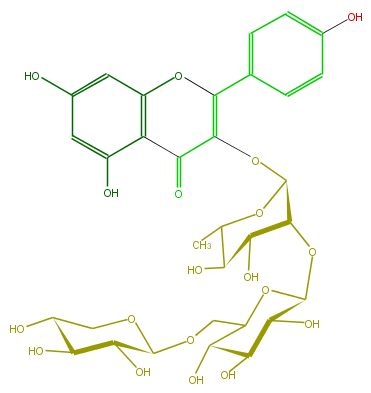
Mol:FL5FAAGS0078
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.2824 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2824 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2824 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2824 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9968 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9968 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7113 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7113 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7113 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7113 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9968 3.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9968 3.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5678 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5678 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1467 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1467 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8612 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8612 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8612 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8612 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1467 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1467 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5678 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5678 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5756 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5756 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2901 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2901 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2901 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2901 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5756 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5756 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1467 0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1467 0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0046 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0046 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3832 0.7272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3832 0.7272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3120 3.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3120 3.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5756 0.0711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5756 0.0711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6956 -0.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6956 -0.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7581 -1.4278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7581 -1.4278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2851 -0.7931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2851 -0.7931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0621 -0.5156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0621 -0.5156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9997 0.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9997 0.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4725 -0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4725 -0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5294 -1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5294 -1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2319 -1.6087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2319 -1.6087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2386 -1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2386 -1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2018 -0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2018 -0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4561 -2.9943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4561 -2.9943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2548 -3.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2548 -3.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6651 -2.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6651 -2.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3827 -2.0779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3827 -2.0779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5840 -1.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5840 -1.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1736 -2.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1736 -2.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5442 -2.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5442 -2.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0929 -2.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0929 -2.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1336 -3.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1336 -3.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7600 -3.5702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7600 -3.5702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4578 -2.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4578 -2.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6738 -2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6738 -2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2613 -3.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2613 -3.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4632 -2.9321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4632 -2.9321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6700 -3.1589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6700 -3.1589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0825 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0825 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8806 -2.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8806 -2.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9290 -3.5033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9290 -3.5033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9308 -3.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9308 -3.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3120 -2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3120 -2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 28 1 0 0 0 0 | + | 35 28 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 43 51 1 0 0 0 0 | + | 43 51 1 0 0 0 0 |
| − | 46 39 1 0 0 0 0 | + | 46 39 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0078 | + | ID FL5FAAGS0078 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES Oc(c6)c(C2=O)c(cc6O)OC(=C2OC(O5)C(C(C(O)C(C)5)O)OC(C4O)OC(C(C4O)O)COC(O3)C(O)C(C(C3)O)O)c(c1)ccc(O)c1 | + | SMILES Oc(c6)c(C2=O)c(cc6O)OC(=C2OC(O5)C(C(C(O)C(C)5)O)OC(C4O)OC(C(C4O)O)COC(O3)C(O)C(C(C3)O)O)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
1.2824 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2824 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9968 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7113 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7113 3.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9968 3.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5678 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1467 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8612 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8612 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1467 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5678 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5756 2.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2901 2.0081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2901 1.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5756 0.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1467 0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0046 2.4206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3832 0.7272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3120 3.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5756 0.0711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6956 -0.6052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7581 -1.4278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2851 -0.7931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0621 -0.5156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9997 0.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4725 -0.3276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5294 -1.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2319 -1.6087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2386 -1.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2018 -0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4561 -2.9943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2548 -3.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6651 -2.4849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3827 -2.0779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5840 -1.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1736 -2.5873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5442 -2.4964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0929 -2.8714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1336 -3.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7600 -3.5702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4578 -2.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6738 -2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2613 -3.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4632 -2.9321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6700 -3.1589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0825 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8806 -2.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9290 -3.5033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9308 -3.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3120 -2.6412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
25 28 1 0 0 0 0
24 29 1 0 0 0 0
23 30 1 0 0 0 0
22 31 1 0 0 0 0
26 19 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 28 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
45 49 1 0 0 0 0
44 50 1 0 0 0 0
43 51 1 0 0 0 0
46 39 1 0 0 0 0
S SKP 5
ID FL5FAAGS0078
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES Oc(c6)c(C2=O)c(cc6O)OC(=C2OC(O5)C(C(C(O)C(C)5)O)OC(C4O)OC(C(C4O)O)COC(O3)C(O)C(C(C3)O)O)c(c1)ccc(O)c1
M END