Mol:FL5FAAGL0053
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8026 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8026 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8026 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8026 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0645 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0645 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3263 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3263 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3263 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3263 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0645 0.3115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0645 0.3115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5882 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5882 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1499 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1499 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1499 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1499 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5882 0.3115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5882 0.3115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5882 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5882 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0793 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0793 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8316 0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8316 0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5839 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5839 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5839 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5839 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8316 1.7786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8316 1.7786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0793 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0793 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0645 -2.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0645 -2.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2873 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2873 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9812 -1.3880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9812 -1.3880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4959 0.3710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4959 0.3710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0575 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0575 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7015 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7015 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3859 -1.4658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3859 -1.4658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0746 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0746 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4306 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4306 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7461 -1.2405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7461 -1.2405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4188 -1.0831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4188 -1.0831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2503 -0.7010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2503 -0.7010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0227 -1.0981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0227 -1.0981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9204 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9204 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2235 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2235 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4447 1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4447 1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2235 1.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2235 1.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9204 2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9204 2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6993 1.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6993 1.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5941 2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5941 2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1998 2.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1998 2.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3867 1.9699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3867 1.9699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5283 0.6802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5283 0.6802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6097 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6097 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4299 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4299 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8403 1.5267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8403 1.5267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6389 2.3266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6389 2.3266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8185 2.3267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8185 2.3267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4080 1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4080 1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3770 1.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3770 1.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9250 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9250 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5283 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5283 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5086 1.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5086 1.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8807 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8807 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8524 -2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8524 -2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | 32 21 1 0 0 0 0 | + | 32 21 1 0 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 41 1 1 0 0 0 | + | 46 41 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 41 48 1 0 0 0 0 | + | 41 48 1 0 0 0 0 |
| − | 42 49 1 0 0 0 0 | + | 42 49 1 0 0 0 0 |
| − | 43 50 1 0 0 0 0 | + | 43 50 1 0 0 0 0 |
| − | 19 45 1 0 0 0 0 | + | 19 45 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 25 51 1 0 0 0 0 | + | 25 51 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 57 -0.8061 -0.1153 | + | M SBV 1 57 -0.8061 -0.1153 |
| − | S SKP 5 | + | S SKP 5 |
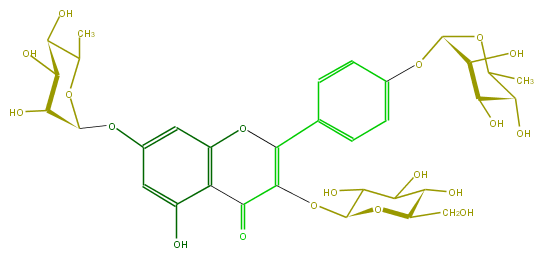
| − | ID FL5FAAGL0053 | + | ID FL5FAAGL0053 |
| − | FORMULA C33H40O19 | + | FORMULA C33H40O19 |
| − | EXACTMASS 740.216379098 | + | EXACTMASS 740.216379098 |
| − | AVERAGEMASS 740.6593 | + | AVERAGEMASS 740.6593 |
| − | SMILES C(C1O)(O)C(CO)OC(OC(C2=O)=C(c(c5)ccc(OC(C(O)6)OC(C)C(O)C6O)c5)Oc(c3)c2c(O)cc(OC(C4O)OC(C)C(C4O)O)3)C1O | + | SMILES C(C1O)(O)C(CO)OC(OC(C2=O)=C(c(c5)ccc(OC(C(O)6)OC(C)C(O)C6O)c5)Oc(c3)c2c(O)cc(OC(C4O)OC(C)C(C4O)O)3)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-2.8026 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8026 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0645 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3263 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3263 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0645 0.3115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5882 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1499 -0.9670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1499 -0.1146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5882 0.3115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5882 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0793 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8316 0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5839 0.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5839 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8316 1.7786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0793 1.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0645 -2.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2873 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9812 -1.3880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4959 0.3710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0575 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7015 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3859 -1.4658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0746 -1.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4306 -1.0449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7461 -1.2405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4188 -1.0831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2503 -0.7010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0227 -1.0981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9204 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2235 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4447 1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2235 1.8447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9204 2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6993 1.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5941 2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1998 2.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3867 1.9699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5283 0.6802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6097 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4299 0.9563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8403 1.5267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6389 2.3266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8185 2.3267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4080 1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3770 1.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9250 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5283 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5086 1.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8807 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8524 -2.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 20 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
36 39 1 0 0 0 0
31 40 1 0 0 0 0
32 21 1 0 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 41 1 1 0 0 0
46 47 1 0 0 0 0
41 48 1 0 0 0 0
42 49 1 0 0 0 0
43 50 1 0 0 0 0
19 45 1 0 0 0 0
51 52 1 0 0 0 0
25 51 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 CH2OH
M SBV 1 57 -0.8061 -0.1153
S SKP 5
ID FL5FAAGL0053
FORMULA C33H40O19
EXACTMASS 740.216379098
AVERAGEMASS 740.6593
SMILES C(C1O)(O)C(CO)OC(OC(C2=O)=C(c(c5)ccc(OC(C(O)6)OC(C)C(O)C6O)c5)Oc(c3)c2c(O)cc(OC(C4O)OC(C)C(C4O)O)3)C1O
M END