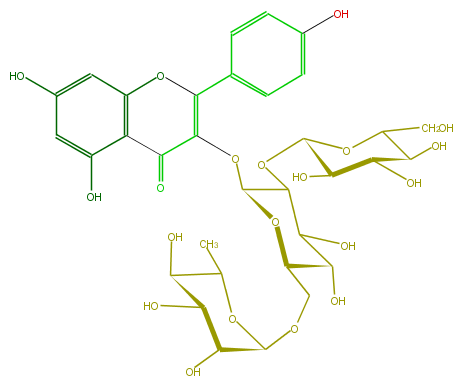
Mol:FL5FAAGL0030
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4495 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4495 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4495 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4495 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0475 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0475 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0475 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0475 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 2.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 2.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3465 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3465 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6454 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6454 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6454 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6454 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3465 2.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3465 2.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3465 0.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3465 0.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0553 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0553 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7698 1.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7698 1.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4842 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4842 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4842 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4842 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7698 3.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7698 3.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0553 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0553 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 -0.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 -0.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1503 2.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1503 2.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1985 3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1985 3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1413 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1413 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1655 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1655 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0797 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0797 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4226 -0.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4226 -0.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1981 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1981 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2839 0.0482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2839 0.0482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9409 -0.5876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9409 -0.5876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3232 -1.0510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3232 -1.0510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1265 -2.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1265 -2.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6607 0.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6607 0.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6164 -2.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6164 -2.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5074 1.0721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5074 1.0721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0649 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0649 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8677 0.6484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8677 0.6484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6423 0.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6423 0.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0794 1.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0794 1.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3772 0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3772 0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5067 0.3132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5067 0.3132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6513 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6513 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1503 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1503 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1704 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1704 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7437 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7437 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0866 -2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0866 -2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1379 -1.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1379 -1.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1560 -1.6775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1560 -1.6775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4991 -2.3132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4991 -2.3132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1340 -0.8768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1340 -0.8768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4943 -1.0100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4943 -1.0100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4711 -2.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4711 -2.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7618 -3.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7618 -3.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3367 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3367 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9315 1.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9315 1.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6953 2.5373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6953 2.5373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 41 1 1 0 0 0 | + | 46 41 1 1 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 46 49 1 0 0 0 0 | + | 46 49 1 0 0 0 0 |
| − | 41 50 1 0 0 0 0 | + | 41 50 1 0 0 0 0 |
| − | 42 51 1 0 0 0 0 | + | 42 51 1 0 0 0 0 |
| − | 51 31 1 0 0 0 0 | + | 51 31 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 36 52 1 0 0 0 0 | + | 36 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 58 -0.8521 -0.0796 | + | M SBV 1 58 -0.8521 -0.0796 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0030 | + | ID FL5FAAGL0030 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES OC(C(OC(C6O)OC(CO)C(C(O)6)O)1)C(O)C(COC(C(O)5)OC(C)C(O)C(O)5)OC1OC(C(=O)2)=C(c(c4)ccc(O)c4)Oc(c3)c2c(O)cc3O | + | SMILES OC(C(OC(C6O)OC(CO)C(C(O)6)O)1)C(O)C(COC(C(O)5)OC(C)C(O)C(O)5)OC1OC(C(=O)2)=C(c(c4)ccc(O)c4)Oc(c3)c2c(O)cc3O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-3.4495 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4495 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0475 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0475 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 2.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3465 0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6454 1.1302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6454 1.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3465 2.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3465 0.0943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0553 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7698 1.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4842 2.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4842 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7698 3.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0553 3.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 -0.0837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1503 2.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1985 3.5815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1413 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1655 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0797 -1.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4226 -0.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1981 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2839 0.0482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9409 -0.5876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3232 -1.0510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1265 -2.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6607 0.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6164 -2.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5074 1.0721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0649 0.3363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8677 0.6484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6423 0.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0794 1.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3772 0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5067 0.3132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6513 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1503 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1704 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7437 -3.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0866 -2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1379 -1.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1560 -1.6775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4991 -2.3132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1340 -0.8768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4943 -1.0100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4711 -2.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7618 -3.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3367 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9315 1.2994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6953 2.5373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
26 21 1 0 0 0 0
25 30 1 0 0 0 0
22 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
30 32 1 0 0 0 0
35 40 1 0 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 41 1 1 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
46 49 1 0 0 0 0
41 50 1 0 0 0 0
42 51 1 0 0 0 0
51 31 1 0 0 0 0
52 53 1 0 0 0 0
36 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 CH2OH
M SBV 1 58 -0.8521 -0.0796
S SKP 5
ID FL5FAAGL0030
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES OC(C(OC(C6O)OC(CO)C(C(O)6)O)1)C(O)C(COC(C(O)5)OC(C)C(O)C(O)5)OC1OC(C(=O)2)=C(c(c4)ccc(O)c4)Oc(c3)c2c(O)cc3O
M END