Mol:FL5FAAGI0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6362 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6362 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6362 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6362 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5236 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5236 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5236 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5236 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9673 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9673 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4110 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4110 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4110 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4110 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9673 1.0627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9673 1.0627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9673 -0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9673 -0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1451 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1451 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7121 0.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7121 0.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2791 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2791 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2791 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2791 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7121 2.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7121 2.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1451 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1451 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1923 1.0626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1923 1.0626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8459 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8459 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0168 -0.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0168 -0.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 -0.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 -0.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 1.7048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 1.7048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6360 2.0259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6360 2.0259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6360 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6360 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1921 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1921 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7307 -1.7235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7307 -1.7235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2661 -2.0326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2661 -2.0326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0962 -1.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0962 -1.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2661 -0.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2661 -0.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7307 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7307 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9005 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9005 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5570 -1.1254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5570 -1.1254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8682 -2.2367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8682 -2.2367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1369 -2.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1369 -2.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6248 -1.7433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6248 -1.7433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2982 -2.1979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2982 -2.1979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8336 -2.5070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8336 -2.5070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6637 -1.9125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6637 -1.9125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8336 -1.3145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8336 -1.3145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2982 -1.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2982 -1.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4680 -1.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4680 -1.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1245 -1.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1245 -1.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4357 -2.7111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4357 -2.7111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7044 -2.9891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7044 -2.9891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1923 -2.2177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1923 -2.2177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 27 1 1 0 0 0 | + | 32 27 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 27 34 1 0 0 0 0 | + | 27 34 1 0 0 0 0 |
| − | 28 35 1 0 0 0 0 | + | 28 35 1 0 0 0 0 |
| − | 29 36 1 0 0 0 0 | + | 29 36 1 0 0 0 0 |
| − | 31 20 1 0 0 0 0 | + | 31 20 1 0 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 37 1 1 0 0 0 | + | 42 37 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 38 45 1 0 0 0 0 | + | 38 45 1 0 0 0 0 |
| − | 39 46 1 0 0 0 0 | + | 39 46 1 0 0 0 0 |
| − | 41 33 1 0 0 0 0 | + | 41 33 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGI0006 | + | ID FL5FAAGI0006 |
| − | KNApSAcK_ID C00005809 | + | KNApSAcK_ID C00005809 |
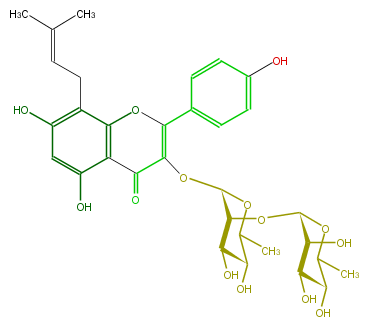
| − | NAME Noranhydroicaritin 3-rhamnosyl-(1->2)-rhamnoside | + | NAME Noranhydroicaritin 3-rhamnosyl-(1->2)-rhamnoside |
| − | CAS_RN 135293-12-8 | + | CAS_RN 135293-12-8 |
| − | FORMULA C32H38O14 | + | FORMULA C32H38O14 |
| − | EXACTMASS 646.226155924 | + | EXACTMASS 646.226155924 |
| − | AVERAGEMASS 646.63572 | + | AVERAGEMASS 646.63572 |
| − | SMILES c(c5O)cc(cc5)C(=C(OC(C3OC(C(O)4)OC(C(O)C4O)C)OC(C(O)C3O)C)2)Oc(c(C(=O)2)1)c(CC=C(C)C)c(O)cc1O | + | SMILES c(c5O)cc(cc5)C(=C(OC(C3OC(C(O)4)OC(C(O)C4O)C)OC(C(O)C3O)C)2)Oc(c(C(=O)2)1)c(CC=C(C)C)c(O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-2.6362 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6362 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5236 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5236 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9673 -0.2220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4110 0.0992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4110 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9673 1.0627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9673 -0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1451 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7121 0.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2791 1.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2791 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7121 2.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1451 1.7173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1923 1.0626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8459 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0168 -0.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 -0.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 1.7048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6360 2.0259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6360 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1921 2.9891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7307 -1.7235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2661 -2.0326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0962 -1.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2661 -0.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7307 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9005 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5570 -1.1254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8682 -2.2367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1369 -2.5147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6248 -1.7433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2982 -2.1979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8336 -2.5070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6637 -1.9125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8336 -1.3145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2982 -1.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4680 -1.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1245 -1.5998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4357 -2.7111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7044 -2.9891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1923 -2.2177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
6 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
24 26 1 0 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 27 1 1 0 0 0
32 33 1 0 0 0 0
27 34 1 0 0 0 0
28 35 1 0 0 0 0
29 36 1 0 0 0 0
31 20 1 0 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
42 37 1 1 0 0 0
42 43 1 0 0 0 0
37 44 1 0 0 0 0
38 45 1 0 0 0 0
39 46 1 0 0 0 0
41 33 1 0 0 0 0
S SKP 8
ID FL5FAAGI0006
KNApSAcK_ID C00005809
NAME Noranhydroicaritin 3-rhamnosyl-(1->2)-rhamnoside
CAS_RN 135293-12-8
FORMULA C32H38O14
EXACTMASS 646.226155924
AVERAGEMASS 646.63572
SMILES c(c5O)cc(cc5)C(=C(OC(C3OC(C(O)4)OC(C(O)C4O)C)OC(C(O)C3O)C)2)Oc(c(C(=O)2)1)c(CC=C(C)C)c(O)cc1O
M END