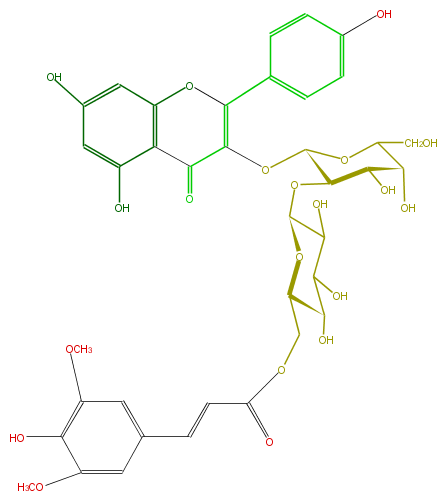
Mol:FL5FAAGA0033
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1005 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1005 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1005 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1005 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3860 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3860 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6716 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6716 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6716 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6716 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3860 3.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3860 3.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9571 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9571 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2427 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2427 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2427 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2427 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9571 3.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9571 3.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9571 1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9571 1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6570 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6570 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3852 3.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3852 3.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1133 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1133 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1133 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1133 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3852 5.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3852 5.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6570 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6570 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3860 1.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3860 1.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8927 5.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8927 5.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7627 3.7642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7627 3.7642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5657 1.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5657 1.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3617 2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3617 2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8776 1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8776 1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6204 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6204 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3373 1.9044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3373 1.9044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8164 2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8164 2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1664 2.0824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1664 2.0824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1585 1.5867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1585 1.5867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9664 1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9664 1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3763 1.1280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3763 1.1280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7482 0.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7482 0.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0377 0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0377 0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2631 0.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2631 0.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0377 -0.6510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0377 -0.6510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7482 -1.0612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7482 -1.0612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5228 -0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5228 -0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6073 1.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6073 1.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0044 -0.6604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0044 -0.6604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7201 -1.5433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7201 -1.5433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2476 -1.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2476 -1.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8934 -2.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8934 -2.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2086 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2086 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6563 -3.6026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6563 -3.6026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5853 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5853 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9788 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9788 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9104 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9104 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3205 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3205 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1406 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1406 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5508 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5508 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1406 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1406 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3205 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3205 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3702 -3.5086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3702 -3.5086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4526 2.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4526 2.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3702 1.9085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3702 1.9085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3895 -1.7198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3895 -1.7198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4719 -2.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4719 -2.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9793 -4.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9793 -4.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0617 -5.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0617 -5.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 26 53 1 0 0 0 0 | + | 26 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 48 55 1 0 0 0 0 | + | 48 55 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 50 57 1 0 0 0 0 | + | 50 57 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 59 -0.6362 -0.0128 | + | M SBV 1 59 -0.6362 -0.0128 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 55 56 | + | M SAL 2 2 55 56 |
| − | M SBL 2 1 61 | + | M SBL 2 1 61 |
| − | M SMT 2 ^OCH3 | + | M SMT 2 ^OCH3 |
| − | M SBV 2 61 0.2489 -1.0786 | + | M SBV 2 61 0.2489 -1.0786 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 57 58 | + | M SAL 3 2 57 58 |
| − | M SBL 3 1 63 | + | M SBL 3 1 63 |
| − | M SMT 3 ^OCH3 | + | M SMT 3 ^OCH3 |
| − | M SBV 3 63 0.8387 0.2947 | + | M SBV 3 63 0.8387 0.2947 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGA0033 | + | ID FL5FAAGA0033 |
| − | FORMULA C38H40O20 | + | FORMULA C38H40O20 |
| − | EXACTMASS 816.21129372 | + | EXACTMASS 816.21129372 |
| − | AVERAGEMASS 816.7121999999999 | + | AVERAGEMASS 816.7121999999999 |
| − | SMILES C(C(OC(O5)C(O)C(C(O)C5COC(C=Cc(c6)cc(c(c(OC)6)O)OC)=O)O)1)(O)C(C(OC1OC(C3=O)=C(Oc(c4)c3c(O)cc4O)c(c2)ccc(O)c2)CO)O | + | SMILES C(C(OC(O5)C(O)C(C(O)C5COC(C=Cc(c6)cc(c(c(OC)6)O)OC)=O)O)1)(O)C(C(OC1OC(C3=O)=C(Oc(c4)c3c(O)cc4O)c(c2)ccc(O)c2)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
-3.1005 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1005 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3860 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6716 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6716 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3860 3.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9571 1.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2427 2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2427 3.1815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9571 3.5939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9571 1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6570 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3852 3.3323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1133 3.7527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1133 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3852 5.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6570 4.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3860 1.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8927 5.0435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7627 3.7642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5657 1.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3617 2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8776 1.6077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6204 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3373 1.9044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8164 2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1664 2.0824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1585 1.5867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9664 1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3763 1.1280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7482 0.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0377 0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2631 0.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0377 -0.6510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7482 -1.0612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5228 -0.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6073 1.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0044 -0.6604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7201 -1.5433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2476 -1.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8934 -2.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2086 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6563 -3.6026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5853 -2.8271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9788 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9104 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3205 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1406 -2.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5508 -3.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1406 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3205 -4.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3702 -3.5086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4526 2.4382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3702 1.9085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3895 -1.7198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4719 -2.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9793 -4.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0617 -5.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 22 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
34 40 1 0 0 0 0
32 28 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 46 1 0 0 0 0
49 52 1 0 0 0 0
53 54 1 0 0 0 0
26 53 1 0 0 0 0
55 56 1 0 0 0 0
48 55 1 0 0 0 0
57 58 1 0 0 0 0
50 57 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 CH2OH
M SBV 1 59 -0.6362 -0.0128
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 55 56
M SBL 2 1 61
M SMT 2 ^OCH3
M SBV 2 61 0.2489 -1.0786
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 57 58
M SBL 3 1 63
M SMT 3 ^OCH3
M SBV 3 63 0.8387 0.2947
S SKP 5
ID FL5FAAGA0033
FORMULA C38H40O20
EXACTMASS 816.21129372
AVERAGEMASS 816.7121999999999
SMILES C(C(OC(O5)C(O)C(C(O)C5COC(C=Cc(c6)cc(c(c(OC)6)O)OC)=O)O)1)(O)C(C(OC1OC(C3=O)=C(Oc(c4)c3c(O)cc4O)c(c2)ccc(O)c2)CO)O
M END